更新: 2018-01-16 12:26:08
生態学のデータ解析 - coda 雑
- ここでは R の package のひとつ
library(coda)を簡単に紹介します- R の help: library(coda)
-
library(coda)は MCMC 計算の出力 (CODA 書式) を R で処理するための package です
- 参照: ベイズ統計 & MCMC, JAGS 雑, R2WinBUGS
[もくじ]
MCMC 計算出力を読みこむ
- JAGS の出力結果をよみこむ:
read.jags()関数 - 読みこんだ内容は
mcmcクラスのオブジェクトになる
図の出力
- ある
mcmcクラスオブジェクトをmとしよう -
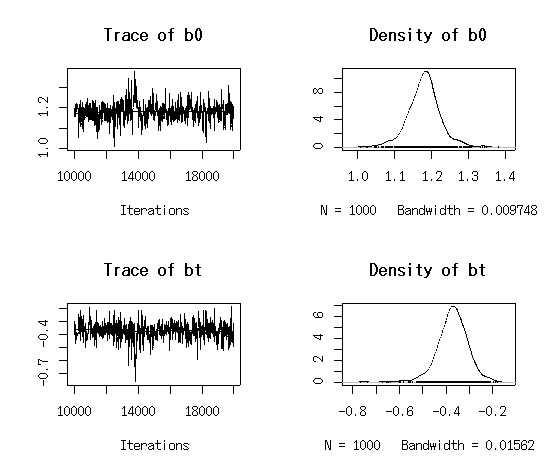
plot(m)で全パラメーターの MCMC 計算遷移図と事後分布の密度関数図が出力される (plot.mcmc()がよびだされている)- それぞれ
traceplot(),densplot()で個別に描ける -
plot.mcmc()の出力例:
- それぞれ
chain ごとに計算遷移図をだす
-
mcmcオブジェクトではなくmcmc.listオブジェクトに MCMC 計算の結果を (chain ごとに) 格納している場合にplot()すると chain ごとの計算遷移がわかる (2006-12-06 遠洋水研の岡村寛さんに教えていただいた).
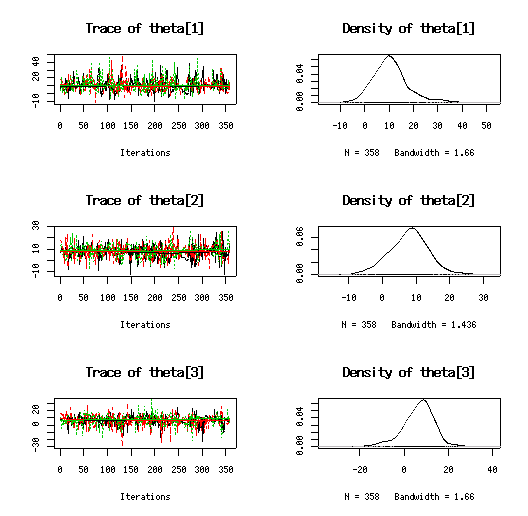
表の出力
- ある
mcmcクラスオブジェクトをmとしよう -
summary(m)で全パラメーターの事後分布の平均・標準偏差,{2.5, 25, 50, 75, 97.5}% 分位点の表が出力される - 出力例:
1. Empirical mean and standard deviation for each variable, plus standard error of the mean: Mean SD Naive SE Time-series SE b0 1.5707 0.186 0.00587 0.01693 bt 0.3129 0.857 0.02709 0.11263 a0 0.2603 0.124 0.00391 0.00788 at -0.5195 0.173 0.00546 0.01491 iv.x 68.4803 279.713 8.84529 17.38780 iv.g 65.0620 249.301 7.88358 13.45898 iv.a 0.0832 0.572 0.01810 0.02830 2. Quantiles for each variable: 2.5% 25% 50% 75% 97.5% b0 1.20958 1.4580 1.603 1.6914 1.861 bt -0.78595 -0.2827 0.167 0.6841 2.914 a0 0.00788 0.1910 0.260 0.3308 0.512 at -0.87161 -0.6331 -0.517 -0.4075 -0.187 iv.x 0.06327 0.2717 0.597 3.8881 791.402 iv.g 0.00583 0.0205 0.293 15.5221 670.739 iv.a 0.00985 0.0309 0.047 0.0717 0.147
データのクラス変換
- ある
mcmcクラスオブジェクトをmとしよう -
as.matrix(m),as.data.frame(m)でそれぞれmatrix,data.frameクラスのオブジェクトに変換される