更新: 2012-09-24 21:50:27
生態学のデータ解析 - lmer MCMC 計算
- cf. lmer 紹介, GLMM 参照, ベイズ統計 & MCMC, coda 雑
- R の help: mcmcsamp(), lmer()
- ここは lmer() (in
library(lme4)) が生成する線形混合モデル (lmer)・一般化線形混合モデル (glmer) オブジェクトを階層ベイズモデルとみなして,その MCMC 計算する関数mcmcsamp()(同じくlibrary(lme4)) の説明です. - 現時点の久保の理解: つまり線形混合モデル・一般化線形混合モデルの階層ベイズモデルの MCMC 計算は R2WinBUGS など使わずとも
lmer()とmcmcsamp()使えばできるらしい (もちろん複数の random effects をあつかえる).- fixed effects の事前分布は一様分布 (局所的無情報事前分布)
- random effects の事前分布は (おそらく) 平均ゼロの多変量正規分布で,その分散共分散行列の超事前分布は (逆) Wishart 分布
とりあえず,の例題
library(lme4) の lmer() 関数に一般化線形混合モデルの問題を解かせると
fit1 <- lmer(y ~ (1 | group), family = binomial, method = "Laplace", ...)
glmer オブジェクト fit1 が作られる.
これを summary(fit1) すると……
Generalized linear mixed model fit using Laplace
Formula: y ~ (1 | group)
Family: binomial(logit link)
AIC BIC logLik deviance
1385 1395 -690 1381
Random effects:
Groups Name Variance Std.Dev.
group (Intercept) 0.0353 0.188
number of obs: 1000, groups: group, 10
Estimated scale (compare to 1 ) 0.9976
Fixed effects:
Estimate Std. Error z value Pr(>|z|)
(Intercept) 0.108 0.087 1.24 0.22
次に glmer オブジェクト fit1 を mcmcsamp() 関数に渡すと
mcmc1 <- mcmcsamp(fit1, n = 500) # library(coda) の mcmc オブジェクト
mcmc オブジェクト mcmc1 が作られるので
- 注: lme4 version 0.999375-31 の場合,
mcmcオブジェクトではない
(注: library(coda) も忘れず実行してから)
summary(mcmc1) を実行すると
- 注: lme4 version 0.999375-31 の場合,
summary(as.mcmc(as.matrix(mcmc1)))とする必要がある?!
Iterations = 1:500
Thinning interval = 1
Number of chains = 1
Sample size per chain = 500
1. Empirical mean and standard deviation for each variable,
plus standard error of the mean:
Mean SD Naive SE Time-series SE
(Intercept) 0.109 0.0999 0.00447 0.00528
log(grop.(In)) -3.391 1.1070 0.04951 0.14390
deviance 1385.842 6.2637 0.28012 0.47325
2. Quantiles for each variable:
2.5% 25% 50% 75% 97.5%
(Intercept) -0.084 0.0474 0.104 0.169 0.318
log(grop.(In)) -6.022 -3.9808 -3.269 -2.629 -1.521
deviance 1375.724 1381.4101 1385.200 1389.406 1399.566
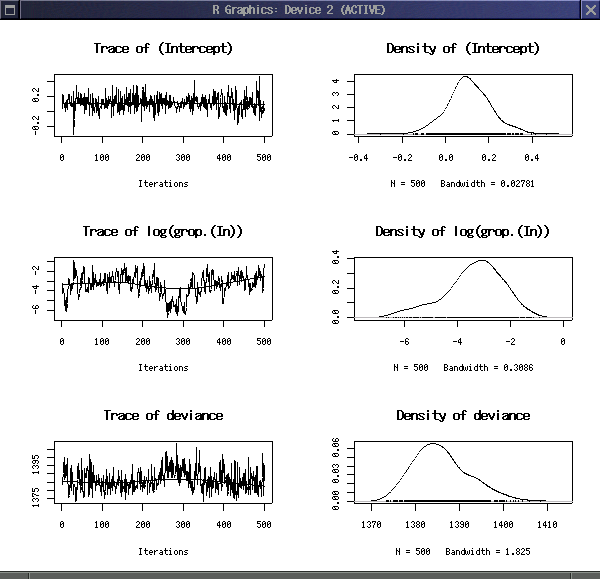
- MCMC 計算の結果に含まれてる
log(grop.(In))はgroupの random effects (つまり group 差) あらわす 事後分布の分散の log になっている -
plot(mcmc1)するといつものごとくこういう図になる- 注: lme4 version 0.999375-31 の場合,
plot(as.mcmc(as.matrix(mcmc1)))とする必要がある?!-
xyplot(mcmc1)だのdensityplot(mcmc1)でよい,とわかりました
-
- 注: lme4 version 0.999375-31 の場合,