更新: 2012-09-24 21:50:27
生態学のデータ解析 - R2WBwrapper
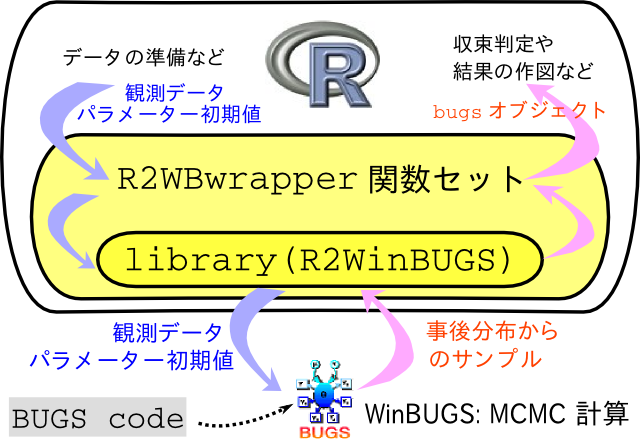
- R2WinBUGS が使いにくいので作った wrapper 関数群についての説明です
- ようするに R から WinBUGS を使うのが少しラクになります
- ファイル: R2WBwrapper.R …… これをダウンロードしてください
-
library(R2WinBUGS)が必要,install.packages("R2WinBUGS")でインストールできます
-
- 参照: R2WinBUGS, 例/car.normal()
- 久保用メモ:
- わたすデータのチェックをする? (data.frame は許可しない,とか)
[もくじ]
使用例
- 例/car.normal() に掲載している
R2WBwrapper.Rの使用例です- 内容は
- データの準備
- パラメーターの初期値の設定 (そして WinBUGS が結果を保存するかどうかの設定)
- WinBUGS の呼び出しとその結果のうけとり
- 内容は
source("R2WBwrapper.R") # R2WBwrapper.R の読みこみ
clear.data.param() # for initialization
# set data (WinBUGS にわたすデータの準備)
load("Y.RData")
set.data("N.site", length(Y))
set.data("Y", Y)
Adj <- c(sapply(2:(N.site - 1), function(a) c(a - 1, a + 1)))
set.data("Adj", c(2, Adj, N.site - 1)) # 「近傍」ID の定義
set.data("Weights", rep(1, 2 * N.site - 2)) # 近傍の重み (全部 1)
set.data("Num", c(1, rep(2, N.site - 2), 1)) # 近傍の個数 (1, 2, 2, ... , 2, 1)
# set parameter (WinBUGS にわたすパラメーター初期値などの準備)
set.param("beta", 0)
set.param("re", rnorm(N.site, 0, 0.1))
set.param("tau", 1)
post.bugs1 <- call.bugs( # ここまで準備したデータとパラメーターをわたして
file.bug = "model1.bug.txt", # model1.bug.txt で定義されているベイズモデルを
n.iter = 200, n.burnin = 100, n.thin = 1 # このような MCMC step で
) # 推定計算して結果を post.bugs1 に格納しろ,という命令
関数の説明
- (あとで書きます)