
[ぎょーむ遂行写真]
生態学会新潟大会,
論文賞受賞式.
左が審査委員長代理の矢原さんが説明してるところ,
右が (海外出張中の) 浦口さん名代の私が賞状うけとっておじぎしてるところ.
はい,
きちんと名代ぎょーむ遂行しました,
ということで.

glmmPQL()
がやたらと使われつつある,
ということ.
こんなの使うんだったらむしろ lmer()
in library(Matrix)
を method = "Laplace"
(すなわち近似的尤度計算)
で使うほうがよほどマシなんぢゃない,
ということで調べてみる
……
やはりそういう結果が得られた.
これは明日にでも解説
(4/2 追記:
ごく簡単な
lmer 紹介
アップロードしました).
よくつかわれてるというコト.glmmPQL(...)よりlmer(..., method = "Laplace")のほうがかなりマシではないでしょうか
rpois()) を使ってゼロに近いところで
「処理」ごとの標本セットを発生,
この状況において変数変換して正規分布で検定
vs glm(y ~ treatment, family = poisson)
で「処理の効果」についてほぼ確実に結果がひっくりかえる事実
(正規分布モデルではゆーい差ナシつまり第二種過誤で
glm()だとアリ)
を示していく
……
ほーら,
なんべんサンプルを生成させても,
と.
つまり「ゆーい差決戦主義」
とやらでポアソン分布モデルの「良さ」を示してるわけね.
lm() と glm()
のつながりを突然にして理解.
とりあえずは,
めでたい.
本日はここまで.
lmer(y ~ (1 | id1) + (1 | id2) + x, data = df, ...)いや, まてよケンカ解析の場合は「random effects の差」が必要なわけだよな, とこれも試してみると,
lmer(y ~ (1 | id1) - (1 | id2) + x, data = df, ...)私が作った簡単な乱数データ例ではかなり正しく計算しているように見える. ふーむ, すごい. (後記: これはたぶんまずいやりかた. こういうふうに単純に
id1
と id2 としてしまうと,
ケンカに勝つときと負けるときで「個体差」 random effects がちがってくる,
といった奇妙なことになってしまいそう).
lmer()
で対戦者それぞれの「個体差」考慮した推定計算できるかもということだ.
しかも method = "Laplace"
で尤度近似計算がうまくいってる,
とみなすなら AIC によるモデル選択
(あるいは尤度比検定など)
もできる.
zicounts()
でやってみたら何やら推定計算がうまくいかなかった
(推定値が NA とかになってしまった)
らしい.
"log"
の場合なんかは悲惨なものになる.
BA を sqrt(BA)
とすればよい.
パラメーター推定の収束計算,
これで何の問題もなくできるようになる
……
といった内容のメイルを,
遅れのおわびとともに吉田さんにおくる.
lmer()
(in library(Matrix))
いいんじゃないの,
と近ごろ私は
宣伝
してるわけだが
……
パラメーター推定計算の収束がうまくいかず,
optim or nlminb returned message singular convergence (7) in: LMEopt(x = mer, value = cv)
といったエラーがでることある.
lmer()
その他の GLMM 推定計算関数で仮定されていることである)
lmer()
というより統計モデルもしくはデータがよくない,
という状況だよね.
lmer()
がオチることがある.
それも R
を道づれにして
……
同様の現象に遭遇した,
と
山口さん
からも教えていただいた.
lmer-class
オブジェクトには事後分布生成のため
mcmcsamp()
という関数が準備されている,
と教えていただく
……
ただしいまこれが適用できるのは非 GLMM,
つまり正規分布の混合モデルの場合だけのようだ.
たしかに random effects がちょっと複雑になると,
事後分布の生成もまた MCMC 計算になっちまうんだねえ
……
readShapePoly()
関数など使うと簡単に ArcGIS 出力ファイルを R の中にとりこむことができる.

plot(obj.SpatialPolygonsDataFrame)]
SpatialPolygonsDataFrame
クラスのオブジェクトになる.
ここから樹冠のカタチのデータであるポリゴンデータをどうとりだすか?
……
いろいろ調べてわかったんだけど,
これは @polygons
スロット (slot) を見ればよい.
スロットについては間瀬さんが
RjpWiki
内に書いておられる
S4 クラスとメソッド入門
を参照.

lmer()
みたいな GLMM 推定関数でなんとかなるかも
……
と書いてたわけだが,
(例によって,というか)
いまごろになってハナシはそう単純ではないかも,
という気がしてきた.
定式化としてはそのときに書いたとおりで問題ない.
しかし,
このまま
lmer()
にほうりこんだりすると,
勝ったときと負けたときで「個体差」 random effects
の大きさが違ってくるはず
……
これはヘンだよね.
やはりできあいの関数では推定計算できず,
自分で尤度方程式書いて
optim()
とかで対数尤度最大化するしかないのか?
data.frame()
つくりなおすという問題.
たとえば
c(2, 1) を c(1, 1, 0)
とするような
……
> test1g x noresp resp total ida 1 1 4 1 5 1 2 2 3 2 5 2 3 3 4 1 5 3 4 3 1 4 5 4 5 4 2 3 5 5 6 5 1 4 5 6 というデータフレームがもとです。説明変数が1のときに反応個体数が1で非反応個体 数が4だった、という内容です。ここからtest1gyという以下のものを作ります。 > test1gy x noresp resp total ida 1 1 0 1 5 1 2 1 1 0 5 1 3 1 1 0 5 1 4 1 1 0 5 1 5 1 1 0 5 1 6 2 0 1 5 2 7 2 0 1 5 2 8 2 1 0 5 2 9 2 1 0 5 2 10 2 1 0 5 2 ... (以下略)
family = binomial)
のパラメーター推定計算やってくれないためだ.
他の GLMM 計算関数
だと cbind(resp, noresp) ~ ...
と書けるのだが.
for (...) { ... }
をたくさん使ったものだったので,
そうではない別の例を作ってみた.
私の返信
(以下で使ってるファイル;
conv01.R,
test1g.csv).
久保です.R ふうコードというべきかどうかわかりませんが,極端な一例とし
て for を使わない変換関数を作ってみました.
# ------------------------------------------------------------------
conv01 <- function(data, resp, noresp, col.join = NULL)
{
# check errors
if (length(resp) != length(noresp))
stop("ERROR: length(resp) != length(noresp)")
if (length(col.join) < 2 & is.null(col.join))
col.join <- colnames(data) # join all columns
# {resp, noresp} --> {0, 1} vector
resp01 <- c(apply(
data.frame(resp, noresp), 1,
function(x) c(rep(1, x[1]), rep(0, x[2]))
))
# join data columns
total <- resp + noresp
cbind(
data.frame("resp01" = resp01), # {0, 1} vector
data[
sapply(1:nrow(data), function(x) rep(x, total[x])),
col.join
]
)
}
# ------------------------------------------------------------------
これを conv01.R といった名前のテキストファイルに保存しておき,
source("conv01.R") とすると conv01.R をよみこんで関数 conv01() の定義
を記憶します.
データが test1g.csv
# ------------------------------------------------------------------
x,noresp,resp,total,ida
1,4,1,5,1
2,3,2,5,2
3,4,1,5,3
3,1,4,5,4
4,2,3,5,5
5,1,4,5,6
# ------------------------------------------------------------------
だとすると
d <- read.csv("test1g.csv")
として
conv01(d, resp = d$resp, noresp = d$noresp)
だの
conv01(d, resp = d$resp, noresp = d$noresp, col.join = c("x", "ida"))
とすれば変換した data.frame を返してきます.
Applied Regression and Multilevel/Hierarchical Models (Analytical Methods for Social Research) (Hardcover) by Andrew Gelman, Jennifer Hill. * Publisher: Cambridge University Press (October 31, 2006) * Language: English * ISBN: 0521867061著者の 本ペイジ に目次などあり.
また、伝票処理についてですが、 調達課から「納品の日付と伝票の日付を同じにする」との通達をいただいております。 発注の手配等をかけることは可能ですが、実際に納品・修理を行うのは、予算が執行されてから、 ということになります。 お待ちいただく形になってしまいますが、ご了承いただきますようよろしくお願いします。予算の執行っていつになるのやら ……
# {resp, noresp} --> {0, 1} vector
resp01 <- c(apply(
data.frame(resp, noresp), 1,
function(x) c(rep(1, x[1]), rep(0, x[2]))
))
とややこしく書いてたけど,
もっと簡単に
resp01 <- rep(c(1, 0), c(resp, noresp))としてよい, とわかった. R ってすごいね.
library(GLMMGibbs)
どうでしょう,
というご質問いただいたのだが
……
これって開発が 2001 年にとまっていて,
あまりおススめではないような.
blas
など線形代数ライブラリと
R
(のライブラリ) が必要とわかった.
bugs-examples
動かしてみる.
時間かかる.
コード例 (これは seeds.bug)
はこんなかんぢで見た目は BUGS 互換.
何やら混合 logistic モデルになっている.
model {
alpha0 ~ dnorm(0.0,1.0E-6); # intercept
alpha1 ~ dnorm(0.0,1.0E-6); # seed coeff
alpha2 ~ dnorm(0.0,1.0E-6); # extract coeff
alpha12 ~ dnorm(0.0,1.0E-6);
tau ~ dgamma(1.0E-3,1.0E-3); # 1/sigma^2
sigma <- 1.0/sqrt(tau);
for (i in 1:N) {
b[i] ~ dnorm(0.0,tau);
logit(p[i]) <- alpha0 + alpha1*x1[i] + alpha2*x2[i] +
alpha12*x1[i]*x2[i] + b[i];
r[i] ~ dbin(p[i],n[i]);
}
}
これを駆動する cmd ファイルはこういうもの.
/* SEEDS example with random effects constrained to sum to zero */ model in "seedszro.bug" data in "seeds-data.R" compile inits in "seeds-init.R" initialize update 1000 monitor set alpha0 , thin(10) monitor set alpha1 , thin(10) monitor set alpha2 , thin(10) monitor set alpha12 , thin(10) monitor set sigma , thin(10) update 40000 coda * exitデータファイルや
"N" <- 21 "r" <- c(10, 23, 23, 26, 17, 5, 53, 55, 32, 46, 10, 8, 10, 8, 23, 0, 3, 22, 15, 32, 3) "n" <- c(39, 62, 81, 51, 39, 6, 74, 72, 51, 79, 13, 16, 30, 28, 45, 4, 12, 41, 30, 51, 7) "x1" <- c(0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1) "x2" <- c(0, 0, 0, 0, 0, 1, 1, 1, 1, 1, 1, 0, 0, 0, 0, 0, 1, 1, 1, 1, 1)パラメーター初期化ファイルは R コードで書かれている.
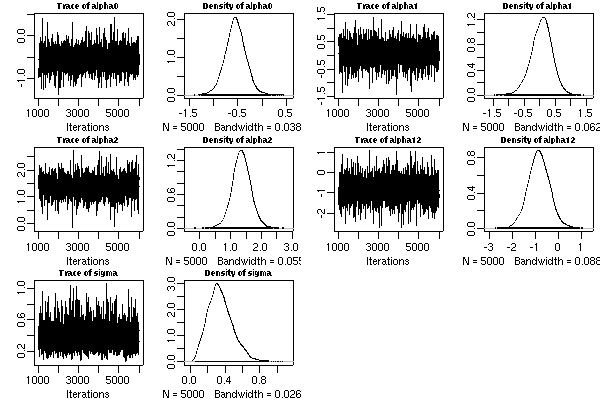
"tau" <- 1 "alpha0" <- 0 "alpha1" <- 0 "alpha2" <- 0 "alpha12" <- 0このように定義されたベイズモデル (混合 logistic モデル) を MCMC 計算させて (
jags cmdファイル名),
その JAGS 出力を library(coda)
の read.jags()
で読んで plot(mcmc(...))
するとこうなる.
gene.str <- list(c("A1", "A2"), c("B1", "B2"))
遺伝子座 A, B
がある,
という状況.
> data A1 A2 B1 B2 100 110 50 65というカタチで格納されていたとする. これを
list()
つかったデータ型に変換する
(ほうがたぶん便利だろうと思ったんで).
> (parent <- get.list.gene(data, gene.str))
[[1]]
A1 A2
100 110
[[2]]
B1 B2
50 65
parent
という名前のおぶぢぇくとにいれてるけど,
これが親だとする.
> get.genotype(parent)
$"A1 A2"
[1] "100/110"
$"B1 B2"
[1] "50/65"
get.genotype()
は単に 100 と 110
をくっつけて 100/110
という遺伝子型にしてるだけである.
parent に自殖
させることにする.
> (possible.child <- parents.to.child(parent, parent))
[[1]]
1 2 3 4
"100/100" "100/110" "100/110" "110/110"
[[2]]
1 2 3 4
"50/50" "50/65" "50/65" "65/65"
これは parent
が自殖したときにできる可能性のある遺伝子型の一覧になっている.
parent
と同じだったとする.
child1 <- parent
parent の遺伝子型がこうなってるから,
> data A1 A2 B1 B2 100 110 50 65子供がこれと同じ遺伝子型である確率は
> get.Mendelian.prob(get.genotype(child1), possible.child) [1] 0.25遺伝子座
A, B
どちらでもヘテロになっている確率なので
0.52 = 0.25 となってるわけだ.
data.frame()
をつっこんだらまとめて list.gene()
に変換してそのリストをかえすような関数が必要だな.
ちなみに入金されているんですか?と聞いたところ「入金がどうこうっていうのは関 係なく、予算執行されていれば大丈夫です!」と言われてしまい、いつ入金されるの かはわかりませんでしたが、とにかく校費での物品購入(発注・納品)はいつでも大 丈夫だそうです。北大生協に注文だす. ついでに何だかよくわからない ETB ユニット, とかいうのも. Canon UM-86E (LBP-2810用 ETBユニット) …… 定着機のことか.
今年度クレーン、ジャングルジム等の利用予定がありましたら、使用者氏名、年齢、 所属、連絡先と使用予定日数、大まかな時期についてお知らせください。これは今月中ぐらいに申請してほしいとのこと. 21 日の苫小牧利用者集合日に申請すればまにあうのではないかしらん …… いや, 私は高所作業とかやらない予定ですけど.
なお、この保険はクレーンだけでなく、ジャングルジムなど苫小牧で行われる高所作 業全般に適用されるものなので、クレーンは使用しなくても高所作業をする予定の方 は必ず申請してください。