コンパイルしてみる.
bugs-examples
動かしてみる.
時間かかる.
コード例 (これは seeds.bug)
はこんなかんぢで見た目は BUGS 互換.
何やら混合 logistic モデルになっている.
model {
alpha0 ~ dnorm(0.0,1.0E-6); # intercept
alpha1 ~ dnorm(0.0,1.0E-6); # seed coeff
alpha2 ~ dnorm(0.0,1.0E-6); # extract coeff
alpha12 ~ dnorm(0.0,1.0E-6);
tau ~ dgamma(1.0E-3,1.0E-3); # 1/sigma^2
sigma <- 1.0/sqrt(tau);
for (i in 1:N) {
b[i] ~ dnorm(0.0,tau);
logit(p[i]) <- alpha0 + alpha1*x1[i] + alpha2*x2[i] +
alpha12*x1[i]*x2[i] + b[i];
r[i] ~ dbin(p[i],n[i]);
}
}
これを駆動する cmd ファイルはこういうもの.
/* SEEDS example with random effects constrained to sum to zero */
model in "seedszro.bug"
data in "seeds-data.R"
compile
inits in "seeds-init.R"
initialize
update 1000
monitor set alpha0 , thin(10)
monitor set alpha1 , thin(10)
monitor set alpha2 , thin(10)
monitor set alpha12 , thin(10)
monitor set sigma , thin(10)
update 40000
coda *
exit
データファイルや
"N" <- 21
"r" <-
c(10, 23, 23, 26, 17, 5, 53, 55, 32, 46, 10, 8, 10, 8, 23, 0,
3, 22, 15, 32, 3)
"n" <-
c(39, 62, 81, 51, 39, 6, 74, 72, 51, 79, 13, 16, 30, 28, 45,
4, 12, 41, 30, 51, 7)
"x1" <-
c(0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1)
"x2" <-
c(0, 0, 0, 0, 0, 1, 1, 1, 1, 1, 1, 0, 0, 0, 0, 0, 1, 1, 1, 1, 1)
パラメーター初期化ファイルは
R コードで書かれている.
"tau" <- 1
"alpha0" <- 0
"alpha1" <- 0
"alpha2" <- 0
"alpha12" <- 0
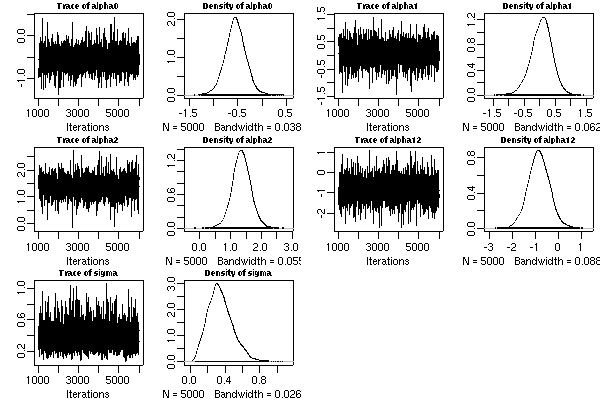
このように定義されたベイズモデル
(混合 logistic モデル)
を MCMC 計算させて
(jags cmdファイル名),
その JAGS 出力を library(coda)
の read.jags()
で読んで plot(mcmc(...))
するとこうなる.