更新: 2012-09-24 21:50:27
生態学のデータ解析 - car.normal() 雑
- WinBUGS の
car.normal()関数のパラメーター設定について,簡単に説明してみます- 参照: 例/car.normal()
[もくじ]
car.normal() の「近傍」設定
- 例/car.normal() の最初の例にそって説明してみます
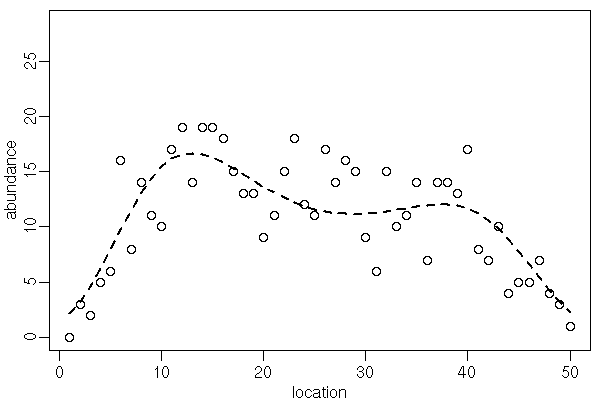
使う例題データ (一次元)
- Y.RData を
load(Y.RData)したときに読みこまれるYは上の図で点々で示されている架空観測値 - 50 ヶ所の観測地は 1, 2, 3, ..., 50 番という「場所 ID」が指定されている (左から 1, 2, 3, ...)
> Y [1] 0 3 2 5 6 16 8 14 11 10 17 19 14 19 19 18 15 13 13 9 11 15 18 12 11 [26] 17 14 16 15 9 6 15 10 11 14 7 14 14 13 17 8 7 10 4 5 5 7 4 3 1
このときの BUGS code
- model1.bug.txt では
Adj,Weights,Numで上のような空間構造つまり「近傍」を指定している- 二次元,三次元といったもっと多次元のデータも同じように「近傍」を指定している
re[1:N.site] ~ car.normal(Adj[], Weights[], Num[], tau) # 場所差を CAR model で生成
「近傍」はどう指定するのか?
-
model1.bug.txtを WinBUGS に実行させる R2WinBUGS な R スクリプト runbugs1 では以下のようにAdj,Weights,Num[]を定義している-
Adj: 「近傍」場所の ID の指定 (長さ 48 * 2 + 2 = 98 の vector) -
Weights: 「近傍」の重み - ここでは全部 1 としている (長さ 98 の vector) -
Num: 「近傍」の個数 (長さ 50 の vector)
-
load("Y.RData")
set.data("N.site", length(Y))
set.data("Y", Y)
Adj <- c(sapply(2:(N.site - 1), function(a) c(a - 1, a + 1)))
set.data("Adj", c(2, Adj, N.site - 1)) # 「近傍」ID の定義
set.data("Weights", rep(1, 2 * N.site - 2)) # 近傍の重み (全部 1)
set.data("Num", c(1, rep(2, N.site - 2), 1)) # 近傍の個数 (1, 2, 2, ... , 2, 1)
具体的に何を指定しているのか
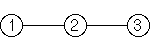
- 50 ヶ所観測値の例を使って説明するのはたいへんなので,観測場所数 が 3 の場合 (上の図) で説明してみます
- 「近傍」場所の ID の指定:
Adj<-c(2,1,3,2)- 以下をくっつけて一次元 vector 化したもの
- 場所 1 の近傍:
2 - 場所 2 の近傍:
1,3 - 場所 3 の近傍:
2
- 場所 1 の近傍:
- 以下をくっつけて一次元 vector 化したもの
- 「近傍」の重み:
Weights<-c(1,1,1,1)- 近傍サイト
Adj<-c(2,1,3,2)の重みがどれも 1 である,と指定している
- 近傍サイト
- 「近傍」の個数:
Num<-c(1,2,1)- 以下をくっつけて一次元 vector 化したもの
- 場所 1 の近傍の個数: 1
- 場所 2 の近傍の個数: 2
- 場所 3 の近傍の個数: 1
- 以下をくっつけて一次元 vector 化したもの
「脱」 car.normal() ?!
-
car.normal()と等価な BUGS code はわりと簡単にかける- こうしたほうがメモリの使用量が少ない,といったハナシがあったりなかったり……