-
岩波本作業のつづき.
りすとらによってとりあえず前後左右がわからなくなったので,
最初の 90 ペイジほどを印刷出力してみることに.
-
Ubuntu になってずいぶんと印刷環境が改善したのだが
……
いろいろと工夫が必要.
-
sudo apt-get install gs-cjk-resource
が必要
(そう,Ubuntu でも GhostScript の呪いからは逃げられない)
-
ちょっとよくわからんのだけど,
たとえば
evince
で印刷出力するときに
too many failed attempts
エラーがでるときに,
「印刷品質」を変更してみるとうまくいくことがあるような気がする
……
ただし
300 dpi
と
600 dpi
のどっちが良いといったハナシではなく,
「変更すること」
自体が何か作用してるようなんだけど
……
わけわかりませんね.
-
ふーむ,
事務室の学術助成係から奇妙なメイルが.
GCOE が吊し上げをくらうのは明後日なのか?
えらくどたばたした対応だな.
あれ?
とっくに終わった 21 世紀 COE とか書いてあるぞ.
あれって何か成果があったのかな.
早速ですが、行政刷新会議対応関係で調査依頼が入りました。
該当事項がある場合は、明日(11/18)14:00までに情報をご提供くださる
よう、よろしくお願いいたします。
【調査内容】
21世紀COEプログラムで育成した博士課程学生、ポスドクのうち、
次のような件があれば、御教示ください。
(1)技術を開発した。もしくは、技術開発のきっかけとなる発見をした。
(2)権威ある賞を獲った。
(3)ベストセラーを執筆した。
(4)有名になった。
(5)その他、成果としてアピールできること。
ポイントは、「一般国民に対し、21世紀COEプログラムからの若手人
材育成の成果として伝わる事例」であること、とのことです。
【提出先】
GCOEユニット
-
生態学会
東京大会
下うけぎょーむ始めなさい,
と鏡味ボスからのご指示が.
203 人分の発表しわけにとりくんでみる.
「しわけ」
は公開するのが近頃の流行のようだから,
公開してみましょう.
-
ここ数年のこのぎょーむの担当者はゑくせるファイル上で
ちこちことひたすら作業してたようだけど,
そんなバカバカしいことはやってられないので
……
とりあえず R でやってみますか.
-
最初のミッション,
1.発表数が2以下の分野は廃止し、第2希望の分野に統合するか、第2希望が廃止分野の場合は第3希望の分野へ統合(ESJ55は、1件のみ第3希望を選択)。
にとりくんでみる.
まずはゑくせる→CSV 化したファイルのよみこみ.
> d0 <- read.csv("ESJ57Oral.csv")
> colnames(d0)
[1] "講演日" "会場" "講演番号" "ユニット" "ユニット内No."
[6] "確定分野" "分野内No." "分野1copy" "分野内No..1" "登録番号"
[11] "名前" "所属" "口頭発表演題" "著者" "分野1"
[16] "分野2" "分野3" "英語可"
「分野 1, 2, 3」だけがとりあえず必要なのだが,
「英語可」もグループわけ (というよりその後の配置) に影響するので,
> d <- d0[, c("分野1", "分野2", "分野3", "英語可")]
としてみる.
-
生態学のデータ解析と同じく,
いろいろと様子をみてみる.
> head(d)
分野1 分野2 分野3 英語可
1 01_群落 02_植物個体群 00_なし -
2 01_群落 11_遷移・更新 00_なし -
3 01_群落 11_遷移・更新 00_なし -
4 01_群落 15_種多様性 00_なし -
5 01_群落 15_種多様性 00_なし -
6 01_群落 24_古生態 00_なし -
> summary(d)
分野1 分野2 分野3 英語可
03_植物生理生態: 16 25_保全 : 24 00_なし :131 - :188
16_数理 : 16 26_生態系管理: 24 26_生態系管理: 9 英語で発表: 15
25_保全 : 15 19_動物個体群: 19 10_景観生態 : 6
26_生態系管理 : 15 17_動物群集 : 12 25_保全 : 6
01_群落 : 13 14_進化 : 11 01_群落 : 5
14_進化 : 12 01_群落 : 10 14_進化 : 5
(Other) :116 (Other) :103 (Other) : 41
-
さてさて,
発表グループ数のうちわけは,
と
……
> g1 <- table(d$分野1) # 発表者数を数えてね
> sort(g1) # 発表者数の少ない順にならべてよ
1 1 1
18_動物繁殖 28_都市 05_植物生活史
1 1 2
12_フェノロジー 13_動物と植物の相互関係 23_分子
2 2 2
29_物質生産 07_種子散布 11_遷移・更新
2 3 3
31_生態学教育・普及 02_植物個体群 04_植物繁殖
3 4 4
15_種多様性 20_動物生活史 22_社会生態
4 5 5
32_英語(分野は不問) 10_景観生態 19_動物個体群
5 7 11
14_進化 17_動物群集 21_行動
12 12 12
27_外来種 30_物質循環 01_群落
12 12 13
25_保全 26_生態系管理 03_植物生理生態
15 15 16
16_数理
16
> g1[g1 < 3] # 発表者数が 3 未満のやつはどれかな?
05_植物生活史 06_送粉 09_微生物
2 1 1
12_フェノロジー 13_動物と植物の相互関係 18_動物繁殖
2 2 1
23_分子 28_都市 29_物質生産
2 1 2
これらのグループは消滅する (第 2 希望以降のグループになる) ことに決定.
-
ちょっとグループ名を確認しておこう.
もともと 32 水準あったわけだが,
08_ と 24_ がなくなったので 30 水準になっている.
> levels.g <- levels(d$分野1)
> levels.g
[1] "01_群落" "02_植物個体群" "03_植物生理生態"
[4] "04_植物繁殖" "05_植物生活史" "06_送粉"
[7] "07_種子散布" "09_微生物" "10_景観生態"
[10] "11_遷移・更新" "12_フェノロジー" "13_動物と植物の相互関係"
[13] "14_進化" "15_種多様性" "16_数理"
[16] "17_動物群集" "18_動物繁殖" "19_動物個体群"
[19] "20_動物生活史" "21_行動" "22_社会生態"
[22] "23_分子" "25_保全" "26_生態系管理"
[25] "27_外来種" "28_都市" "29_物質生産"
[28] "30_物質循環" "31_生態学教育・普及" "32_英語(分野は不問)" :
-
現時点では,
まだどーでもよいのだが,
ちょっとチェックしておくか.
> d[d$分野1 == "32_英語(分野は不問)" & d$英語可 == "英語で発表",]
分野1 分野2 分野3 英語可
199 32_英語(分野は不問) 16_数理 00_なし 英語で発表
200 32_英語(分野は不問) 23_分子 00_なし 英語で発表
201 32_英語(分野は不問) 26_生態系管理 00_なし 英語で発表
202 32_英語(分野は不問) 26_生態系管理 10_景観生態 英語で発表
203 32_英語(分野は不問) 30_物質循環 28_都市 英語で発表
やはり「32_英語(分野は不問)」は全員
「英語可」で動かせるのは 10 名.
しかしこれはあとまわしだ.
-
さーて,
いよいよ「発表者すくなすぎ」発表グループのつけかえ作業だ.
まずは準備として第 2-3 希望の水準をそろえておく.
> d$分野2 <- factor(d$分野2, levels = levels.g)
> d$分野3 <- factor(d$分野3, levels = levels.g)
こうすると,
たとえば,
こんなふうに
sapply()
と
table()
のコンビネイションわざでこんなのができるんだよね.
> sapply(1:3, function(k) table(d[, sprintf("分野%i", k)]))
[,1] [,2] [,3]
01_群落 13 10 5
02_植物個体群 4 6 3
03_植物生理生態 16 3 2
04_植物繁殖 4 6 2
05_植物生活史 2 1 0
06_送粉 1 1 0
07_種子散布 3 0 0
09_微生物 1 2 0
10_景観生態 7 6 6
11_遷移・更新 3 6 0
12_フェノロジー 2 4 1
13_動物と植物の相互関係 2 6 4
14_進化 12 11 5
15_種多様性 4 8 5
16_数理 16 8 2
17_動物群集 12 12 4
18_動物繁殖 1 5 3
19_動物個体群 11 19 4
20_動物生活史 5 8 2
21_行動 12 6 2
22_社会生態 5 4 1
23_分子 2 5 1
25_保全 15 24 6
26_生態系管理 15 24 9
27_外来種 12 3 1
28_都市 1 0 1
29_物質生産 2 6 0
30_物質循環 12 4 0
31_生態学教育・普及 3 0 1
32_英語(分野は不問) 5 3 2
しかしこれは無意味なテイブルだな.
上でやった「levels 変換」はのちのち役にたつのだが.
-
以上のような準備をしておいてから,
> # 発表数 3 未満のグループを廃止
> g <- d$分野1
> table.g <- table(g)
> removed.g1 <- levels.g[table.g < 3]
> removed.g1
[1] "05_植物生活史" "06_送粉" "09_微生物"
[4] "12_フェノロジー" "13_動物と植物の相互関係" "18_動物繁殖"
[7] "23_分子" "28_都市" "29_物質生産"
今度は 203 人の発表者のうち,
以上の発表グループを第一希望にしてるヒトたちは,
と
……
> people.removed.g1 <- g %in% removed.g1
> people.removed.g1 # 203 名のうち第一希望グループで発表できない人
[1] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[15] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[29] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE TRUE TRUE TRUE FALSE FALSE
[43] FALSE TRUE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE TRUE TRUE
[57] TRUE TRUE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[71] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[85] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[99] FALSE FALSE FALSE FALSE TRUE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[113] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[127] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE TRUE TRUE FALSE FALSE
[141] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[155] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[169] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE TRUE TRUE
[183] TRUE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[197] FALSE FALSE FALSE FALSE FALSE FALSE FALSE
> sum(people.removed.g1) # その合計 14 名
[1] 14
> table.g[table.g < 3] # 念のためチェック,たしかに 14 名だ
g
05_植物生活史 06_送粉 09_微生物
2 1 1
12_フェノロジー 13_動物と植物の相互関係 18_動物繁殖
2 2 1
23_分子 28_都市 29_物質生産
2 1 2
> # 第一希望が通らなかった人たちを第二希望に
> g[people.removed.g1] <- d[people.removed.g1, "分野2"]
> sort(table(g))
06_送粉 09_微生物 12_フェノロジー
0 0 0
18_動物繁殖 23_分子 28_都市
0 0 0
29_物質生産 05_植物生活史 13_動物と植物の相互関係
0 1 1
07_種子散布 31_生態学教育・普及 02_植物個体群
3 3 4
11_遷移・更新 15_種多様性 20_動物生活史
4 4 5
32_英語(分野は不問) 04_植物繁殖 22_社会生態
5 6 6
10_景観生態 17_動物群集 19_動物個体群
7 12 12
21_行動 27_外来種 14_進化
12 12 13
30_物質循環 01_群落 16_数理
13 14 16
25_保全 26_生態系管理 03_植物生理生態
16 16 18
う
……
なんじゃこれは
……
ゼロになったグループがあるのはいいとして,
「05_植物生活史」「13_動物と植物の相互関係」
は何で発表者数 1 になるの?
もしや,と思って調べてみたら
……
> d[people.removed.g1,]
分野1 分野2 分野3 英語可
38 05_植物生活史 04_植物繁殖 02_植物個体群 -
39 05_植物生活史 04_植物繁殖 02_植物個体群 -
40 06_送粉 13_動物と植物の相互関係 04_植物繁殖 -
44 09_微生物 26_生態系管理 <NA> -
55 12_フェノロジー 03_植物生理生態 <NA> -
56 12_フェノロジー 11_遷移・更新 03_植物生理生態 -
57 13_動物と植物の相互関係 03_植物生理生態 <NA> -
58 13_動物と植物の相互関係 05_植物生活史 <NA> -
103 18_動物繁殖 22_社会生態 19_動物個体群 -
137 23_分子 14_進化 <NA> -
138 23_分子 19_動物個体群 <NA> -
181 28_都市 25_保全 <NA> 英語で発表
182 29_物質生産 01_群落 <NA> -
183 29_物質生産 30_物質循環 <NA> -
これは第一希望がとおらなかった 14 名のかたですが,
40 06_送粉 13_動物と植物の相互関係 04_植物繁殖 -
とか
58 13_動物と植物の相互関係 05_植物生活史 <NA> -
とか
……
つまり,
第 1 希望も第 2 希望も廃止されちゃうグループだったんですね.
はい.
-
うー,
行名 40 番のヒトは第 3 希望の「04_植物繁殖」にまわってもらうしかないとして
……
行名 58 番のヒトは第 3 希望がない.
えーい,
最初に
CSV そのまま読みこんだ「もと」data.frame
d0
において
d0[58,]
とすると演者・タイトルなどが読めるのですが
(さすがにこの行は「ぎょーむ日誌」で公開できない)
……
なるほど知ってるヒトだ.
まあ,
この内容ならこちらも「04_植物繁殖」
にまわっていただきましょう.
ということで,
> g[40] <- "04_植物繁殖"
> g[58] <- "04_植物繁殖"
> sort(table(g))
g
05_植物生活史 06_送粉 09_微生物
0 0 0
12_フェノロジー 13_動物と植物の相互関係 18_動物繁殖
0 0 0
23_分子 28_都市 29_物質生産
0 0 0
07_種子散布 31_生態学教育・普及 02_植物個体群
3 3 4
11_遷移・更新 15_種多様性 20_動物生活史
4 4 5
32_英語(分野は不問) 22_社会生態 10_景観生態
5 6 7
04_植物繁殖 17_動物群集 19_動物個体群
8 12 12
21_行動 27_外来種 14_進化
12 12 13
30_物質循環 01_群落 16_数理
13 14 16
25_保全 26_生態系管理 03_植物生理生態
16 16 18
これでめでたくどのグループも発表数が 3 以上になった.
-
水準数の再調節.
廃止された発表グループはこの 9 個なのだが,
> table.g <- table(g) # 再計算
> removed.g <- levels.g[table.g == 0]
> removed.g
[1] "05_植物生活史" "06_送粉" "09_微生物"
[4] "12_フェノロジー" "13_動物と植物の相互関係" "18_動物繁殖"
[7] "23_分子" "28_都市" "29_物質生産"
203 人分の確定「発表グループ」の水準つけなおし
(30 水準から上の 9 個を除いた 21 水準)
はこれでよい:
> table(g)
g
01_群落 02_植物個体群 03_植物生理生態 04_植物繁殖
14 4 18 8
07_種子散布 10_景観生態 11_遷移・更新 14_進化
3 7 4 13
15_種多様性 16_数理 17_動物群集 19_動物個体群
4 16 12 12
20_動物生活史 21_行動 22_社会生態 25_保全
5 12 6 16
26_生態系管理 27_外来種 30_物質循環 31_生態学教育・普及
16 12 13 3
32_英語(分野は不問)
5
203 名の発表者の皆さんの発表グループ確定.
めでたしめでたし.
ここまでの作業に使った R コードはこれだけなんだよね.
d0 <- read.csv("ESJ57Oral.csv")
d <- d0[, c("分野1", "分野2", "分野3", "英語可")]
# 水準をそろえる
levels.g <- levels(d$分野1)
d$分野2 <- factor(d$分野2, levels = levels.g)
d$分野3 <- factor(d$分野3, levels = levels.g)
# 発表数 3 未満のグループを廃止
g <- d$分野1
table.g <- table(g)
removed.g1 <- levels.g[table.g < 3]
people.removed.g1 <- g %in% removed.g1
# 第一希望が通らなかった人たちを第二希望に
g[people.removed.g1] <- d[people.removed.g1, "分野2"]
# 第二希望でもダメだった人のグループを手動で操作
g[40] <- "04_植物繁殖"
g[58] <- "04_植物繁殖"
# 発表グループわけ終了,水準数の再調節
table.g <- table(g) # 再計算
removed.g <- levels.g[table.g == 0]
g <- factor(g)
これだけで阿呆らしいゑくせる作業から釈放されます.
-
さて,
次は「どの時間・どの会場に発表グループを配置するか」
問題なんだよね.
いろいろとルールがあるようで
……
2.できるだけ内容の近い分野で、合計してユニット単位の発表数(1件15分、ESJ57東京は1ユニット12件、昨年ESJ56盛岡は、18日の3時間枠で12件・21日の2時間枠で8件)になるような2分野の組み合わせを探す(植物関係、動物関係、物質関係など)。その際、ユニット単位の発表数にそろえるために、若干数の講演は第2希望もしくは第3希望にまわした。
※ESJ57東京では、3時間枠(12件)×18会場(1日当たり9会場、2日間)( 合計216件の枠)。203の申込数をできるだけそのユニットに入るように調整してください。ユニットによっては、11件や8件の発表数になるようにして、203件すべてを割り振ってください。
3.一分野で12件を大きく上回る分野は、2ユニットに分割する。その際、1ユニットは単独分野になるが、もう1ユニットは他の分野との分割になる。どの講演をどちらのユニットに回すかは、講演順番を決めるときにタイトルで判断することも可能。
4.講演数が中途半端な分野がかなり残る場合、2分野を合計してユニット単位の発表数になるような組み合わせを探す。その際、内容の近い組み合わせを優先してつくる。講演数が少ない分野は廃止せず、10以上の講演数をもつ分野と組み合わせる。最後にのこった分野は、内容がかなり違っても、合計がユニット単位の発表数になるような組み合わせをつくる。
めんどくさいね.
-
英語なヒトたちを確認しておくか.
> data.frame(g, d$英語可)[g != "32_英語(分野は不問)" & d$英語可 == "英語で発表",]
g d.英語可
16 02_植物個体群 英語で発表
18 03_植物生理生態 英語で発表
19 03_植物生理生態 英語で発表
20 03_植物生理生態 英語で発表
74 15_種多様性 英語で発表
102 17_動物群集 英語で発表
110 19_動物個体群 英語で発表
168 26_生態系管理 英語で発表
181 25_保全 英語で発表
198 31_生態学教育・普及 英語で発表
まあ,
英語グループに移ってもらうとしたら「03_植物生理生態」
でカタまって,
というかんじかな.
しかしこれはどうしても並びかえがうまくいかなかった場合にしよう.
-
すでに遅くなってきたので本日の作業はここまで.
明日の作業の布石として,
次のような「R コードの部品を R 自身に出力させる」
てなことをやっておいて本日は終了.
> dummy <- sapply(names(table.g), function(k) cat(sprintf("\t\"%s\", # 発表数 %i\n", k, table.g[k])))
"01_群落", # 発表数 14
"02_植物個体群", # 発表数 4
"03_植物生理生態", # 発表数 18
"04_植物繁殖", # 発表数 8
"07_種子散布", # 発表数 3
"10_景観生態", # 発表数 7
"11_遷移・更新", # 発表数 4
"14_進化", # 発表数 13
"15_種多様性", # 発表数 4
"16_数理", # 発表数 16
"17_動物群集", # 発表数 12
"19_動物個体群", # 発表数 12
"20_動物生活史", # 発表数 5
"21_行動", # 発表数 12
"22_社会生態", # 発表数 6
"25_保全", # 発表数 16
"26_生態系管理", # 発表数 16
"27_外来種", # 発表数 12
"30_物質循環", # 発表数 13
"31_生態学教育・普及", # 発表数 3
"32_英語(分野は不問)", # 発表数 5
-
1900 研究室発.
1930 帰宅.
晩飯の準備.
晩飯.
-
夜中ちかくになって,
上で生成した R コード片を
v.g <- c(
"02_植物個体群", # 発表数 4
"03_植物生理生態", # 発表数 18
"SEPARATOR", # 1----------------------------
"01_群落", # 発表数 14
"07_種子散布", # 発表数 3
"10_景観生態", # 発表数 7
"SEPARATOR", # 2----------------------------
"04_植物繁殖", # 発表数 8
"11_遷移・更新", # 発表数 4
"27_外来種", # 発表数 12
"SEPARATOR", # 3----------------------------
"14_進化", # 発表数 13
"15_種多様性", # 発表数 4
"SEPARATOR", # 4----------------------------
"17_動物群集", # 発表数 12
"19_動物個体群", # 発表数 12
"SEPARATOR", # 5----------------------------
"20_動物生活史", # 発表数 5
"21_行動", # 発表数 12
"22_社会生態", # 発表数 6
"SEPARATOR", # 6----------------------------
"16_数理", # 発表数 16
"32_英語(分野は不問)", # 発表数 5
"SEPARATOR", # 7----------------------------
"26_生態系管理", # 発表数 16 (分割前)
"30_物質循環", # 発表数 13
"SEPARATOR", # 8----------------------------
"25_保全", # 発表数 16 (生態系管理分割前)
"31_生態学教育・普及", # 発表数 3
"END" # 9-----------------------------------
)
と vector にしてごちゃごちゃすると,
会場 01 (発表数 22): 02_植物個体群 (4), 03_植物生理生態 (18)
会場 02 (発表数 24): 01_群落 (14), 10_景観生態 (7), 07_種子散布 (3)
会場 03 (発表数 24): 11_遷移・更新 (4), 04_植物繁殖 (8), 27_外来種 (12)
会場 04 (発表数 17): 14_進化 (13), 15_種多様性 (4)
会場 05 (発表数 24): 17_動物群集 (12), 19_動物個体群 (12)
会場 06 (発表数 23): 20_動物生活史 (5), 21_行動 (12), 22_社会生態 (6)
会場 07 (発表数 21): 16_数理 (16), 32_英語(分野は不問) (5)
会場 08 (発表数 24): 26_生態系管理 (11), 30_物質循環 (13)
会場 09 (発表数 24): 25_保全 (21), 31_生態学教育・普及 (3)
とできるとわかった.
-
しかし上の分類は「14_進化 (13)」を含んでいるのでダメ
(とゆールールがある)
と気づいた.
まあ,
試行錯誤は簡単にできるのでまた明日.
-
[今日の運動]
-
[今日の食卓]
- 朝 (0810):
米麦 0.6 合.
ダイコン・ニンジン・ゴボウ・キャベツ・丸天の煮もの.
- 昼 (1340):
北部生協.
自宅からもってきた米麦 0.8 合,
これのおかげで昨日はトラップされてしまった
「寿司」狭隘経路をココロづよく突破することできました!
生協おでん (コンニャク,ロールキャベツ,がんもどき各 52 円)
+ サラダバー.
399 円.
- 晩 (2130):
米麦 0.8 合.
ネギ・シイタケ・春雨・カジカの鍋もの.
ゴボウ・ニンジン・シイタケのゴマ炒め.
-
0720 起床.
ねむい.
背中は痛くはないんだけど,
まだなんか違和感があるんだよね
……
いや,
首を回転させるとまだちょっと痛いかな.
腕立てふせ,
おそるべし.
朝飯.
コーヒー.
0830 自宅発.
曇.
ちょっと雪.
0850 研究室着.
-
生態学会
東京大会
の口頭発表編成の下うけぎょーむのつづき.
えーと
……
とりあえず
問題は「発表数 13」の以下の二つの発表グループを
分割せんといかんあたりで.
なぜかというと 1 日めに 12 個の発表があり,
翌日に 1 つだけの発表になるから.
とりあえず R の data.frame の条件つき表示でながめてみる.
> d[g == "14_進化",]
分野1 分野2 分野3 英語可
59 14_進化 15_種多様性 <NA> -
60 14_進化 16_数理 <NA> -
61 14_進化 17_動物群集 <NA> -
62 14_進化 20_動物生活史 <NA> -
63 14_進化 23_分子 <NA> -
64 14_進化 23_分子 <NA> -
65 14_進化 16_数理 15_種多様性 -
66 14_進化 16_数理 15_種多様性 -
67 14_進化 23_分子 15_種多様性 -
68 14_進化 19_動物個体群 16_数理 -
69 14_進化 21_行動 20_動物生活史 -
70 14_進化 19_動物個体群 23_分子 -
137 23_分子 14_進化 <NA> -
> d[g == "30_物質循環",]
分野1 分野2 分野3 英語可
183 29_物質生産 30_物質循環 <NA> -
184 30_物質循環 09_微生物 <NA> -
185 30_物質循環 10_景観生態 <NA> -
186 30_物質循環 11_遷移・更新 <NA> -
187 30_物質循環 11_遷移・更新 <NA> -
188 30_物質循環 17_動物群集 <NA> -
189 30_物質循環 17_動物群集 <NA> -
190 30_物質循環 25_保全 <NA> -
191 30_物質循環 26_生態系管理 <NA> -
192 30_物質循環 26_生態系管理 <NA> -
193 30_物質循環 26_生態系管理 <NA> -
194 30_物質循環 29_物質生産 <NA> -
195 30_物質循環 17_動物群集 10_景観生態 -
-
じつは昨晩は「生態系管理」を分割していたのだけど,
さらに「進化」「物質循環」も分割してみる.
# 生態系管理の分割 (発表会場の都合により)
g[g == "26_生態系管理" & d$分野2 == "25_保全"] <- "25_保全"
# 「進化」の分割 (発表会場の都合により)
g[g == "14_進化" & d$分野2 == "15_種多様性"] <- "15_種多様性"
# 「物質循環」の分割 (発表会場の都合により)
g[g == "30_物質循環" & d$分野2 == "26_生態系管理"] <- "26_生態系管理"
会場 01 (発表数 22): 02_植物個体群 (4), 03_植物生理生態 (18)
会場 02 (発表数 24): 01_群落 (14), 07_種子散布 (3), 10_景観生態 (7)
会場 03 (発表数 24): 04_植物繁殖 (8), 11_遷移・更新 (4), 27_外来種 (12)
会場 04 (発表数 20): 14_進化 (12), 15_種多様性 (5), 31_生態学教育・普及 (3)
会場 05 (発表数 24): 17_動物群集 (12), 19_動物個体群 (12)
会場 06 (発表数 23): 20_動物生活史 (5), 21_行動 (12), 22_社会生態 (6)
会場 07 (発表数 21): 16_数理 (16), 32_英語(分野は不問) (5)
会場 08 (発表数 24): 26_生態系管理 (14), 30_物質循環 (10)
会場 09 (発表数 21): 25_保全 (21)
-
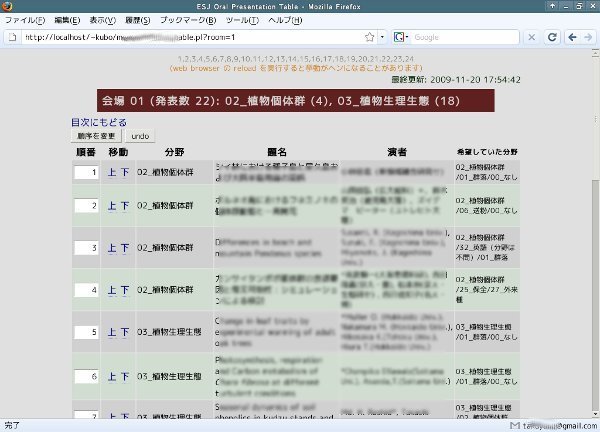
で,
上のような表示は見にくいと編成ボスが困っておられるので,
CSV ファイルな時間割表を作ってみることに.
-
その準備として,
下のようなヘンテコな関数を準備してみる.
# 時間割表生成
n.u <- length(list.u) # ユニット数 (会場数)
n.t <- c(12, 12) # 1 日のコマ数 (12 + 12)
> # 発表時間
> get.ptime <- function(t = 1) {
+ day <- ifelse(t <= n.t[1], "3/15", "3/16")
+ t <- (ifelse(t <= n.t[1], t, t - n.t[1]) - 1) / 4 + 9 # => 9:00-12:00
+ tf <- floor(t) # 1.75 -> 1
+ c(
+ day = day,
+ time = sprintf("%02i:%02i", tf, (t - tf) * 60)
+ )
+ }
> sapply(1:24, get.ptime) # 試験運転
[,1] [,2] [,3] [,4] [,5] [,6] [,7] [,8] [,9] [,10]
day "3/15" "3/15" "3/15" "3/15" "3/15" "3/15" "3/15" "3/15" "3/15" "3/15"
time "09:00" "09:15" "09:30" "09:45" "10:00" "10:15" "10:30" "10:45" "11:00" "11:15"
[,11] [,12] [,13] [,14] [,15] [,16] [,17] [,18] [,19] [,20]
day "3/15" "3/15" "3/16" "3/16" "3/16" "3/16" "3/16" "3/16" "3/16" "3/16"
time "11:30" "11:45" "09:00" "09:15" "09:30" "09:45" "10:00" "10:15" "10:30" "10:45"
[,21] [,22] [,23] [,24]
day "3/16" "3/16" "3/16" "3/16"
time "11:00" "11:15" "11:30" "11:45"
-
で,
こんなかんぢで時間割表生成 & CSV ファイル生成のコードを書いてみる.
# 口頭発表配列
table.ptime <- sapply(1:sum(n.t), get.ptime)
df.p <- data.frame(
day = table.ptime[1,],
time = table.ptime[2,]
)
m.p <- sapply(
1:n.u, function(i) {
unit <- list.u[[i]]
v.p <- rep(unit, table.g[unit])
k <- 0
col.p <- sapply(
1:sum(n.t), function(time) {
k <<- k + 1
v.p[k]
}
)
}
)
df.p <- cbind(df.p, m.p)
file <- "oral.csv"
cat("# output to", file, "...\n")
write.csv(df.p, file = file, row.names = FALSE)
-
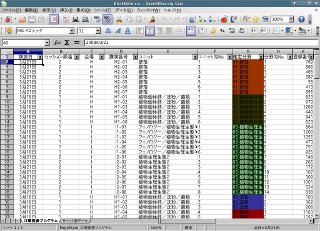
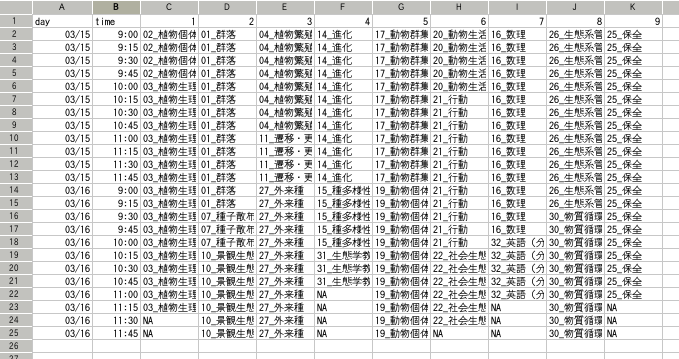
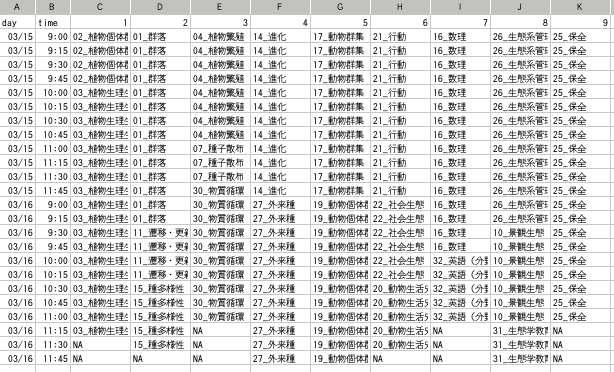
これを実行すると,
CSV ファイル
(注: 編成ボス点検用に Shift-JIS 化されてる)
が生成され,
ゑくせるもしくは
gnumeric
みたいなソフトウェアで開くと,
下の図のようになる.