-
0720 起床.
ねむい.
背中は痛くはないんだけど,
まだなんか違和感があるんだよね
……
いや,
首を回転させるとまだちょっと痛いかな.
腕立てふせ,
おそるべし.
朝飯.
コーヒー.
0830 自宅発.
曇.
ちょっと雪.
0850 研究室着.
-
生態学会
東京大会
の口頭発表編成の下うけぎょーむのつづき.
えーと
……
とりあえず
問題は「発表数 13」の以下の二つの発表グループを
分割せんといかんあたりで.
なぜかというと 1 日めに 12 個の発表があり,
翌日に 1 つだけの発表になるから.
とりあえず R の data.frame の条件つき表示でながめてみる.
> d[g == "14_進化",]
分野1 分野2 分野3 英語可
59 14_進化 15_種多様性 <NA> -
60 14_進化 16_数理 <NA> -
61 14_進化 17_動物群集 <NA> -
62 14_進化 20_動物生活史 <NA> -
63 14_進化 23_分子 <NA> -
64 14_進化 23_分子 <NA> -
65 14_進化 16_数理 15_種多様性 -
66 14_進化 16_数理 15_種多様性 -
67 14_進化 23_分子 15_種多様性 -
68 14_進化 19_動物個体群 16_数理 -
69 14_進化 21_行動 20_動物生活史 -
70 14_進化 19_動物個体群 23_分子 -
137 23_分子 14_進化 <NA> -
> d[g == "30_物質循環",]
分野1 分野2 分野3 英語可
183 29_物質生産 30_物質循環 <NA> -
184 30_物質循環 09_微生物 <NA> -
185 30_物質循環 10_景観生態 <NA> -
186 30_物質循環 11_遷移・更新 <NA> -
187 30_物質循環 11_遷移・更新 <NA> -
188 30_物質循環 17_動物群集 <NA> -
189 30_物質循環 17_動物群集 <NA> -
190 30_物質循環 25_保全 <NA> -
191 30_物質循環 26_生態系管理 <NA> -
192 30_物質循環 26_生態系管理 <NA> -
193 30_物質循環 26_生態系管理 <NA> -
194 30_物質循環 29_物質生産 <NA> -
195 30_物質循環 17_動物群集 10_景観生態 -
-
じつは昨晩は「生態系管理」を分割していたのだけど,
さらに「進化」「物質循環」も分割してみる.
# 生態系管理の分割 (発表会場の都合により)
g[g == "26_生態系管理" & d$分野2 == "25_保全"] <- "25_保全"
# 「進化」の分割 (発表会場の都合により)
g[g == "14_進化" & d$分野2 == "15_種多様性"] <- "15_種多様性"
# 「物質循環」の分割 (発表会場の都合により)
g[g == "30_物質循環" & d$分野2 == "26_生態系管理"] <- "26_生態系管理"
会場 01 (発表数 22): 02_植物個体群 (4), 03_植物生理生態 (18)
会場 02 (発表数 24): 01_群落 (14), 07_種子散布 (3), 10_景観生態 (7)
会場 03 (発表数 24): 04_植物繁殖 (8), 11_遷移・更新 (4), 27_外来種 (12)
会場 04 (発表数 20): 14_進化 (12), 15_種多様性 (5), 31_生態学教育・普及 (3)
会場 05 (発表数 24): 17_動物群集 (12), 19_動物個体群 (12)
会場 06 (発表数 23): 20_動物生活史 (5), 21_行動 (12), 22_社会生態 (6)
会場 07 (発表数 21): 16_数理 (16), 32_英語(分野は不問) (5)
会場 08 (発表数 24): 26_生態系管理 (14), 30_物質循環 (10)
会場 09 (発表数 21): 25_保全 (21)
-
で,
上のような表示は見にくいと編成ボスが困っておられるので,
CSV ファイルな時間割表を作ってみることに.
-
その準備として,
下のようなヘンテコな関数を準備してみる.
# 時間割表生成
n.u <- length(list.u) # ユニット数 (会場数)
n.t <- c(12, 12) # 1 日のコマ数 (12 + 12)
> # 発表時間
> get.ptime <- function(t = 1) {
+ day <- ifelse(t <= n.t[1], "3/15", "3/16")
+ t <- (ifelse(t <= n.t[1], t, t - n.t[1]) - 1) / 4 + 9 # => 9:00-12:00
+ tf <- floor(t) # 1.75 -> 1
+ c(
+ day = day,
+ time = sprintf("%02i:%02i", tf, (t - tf) * 60)
+ )
+ }
> sapply(1:24, get.ptime) # 試験運転
[,1] [,2] [,3] [,4] [,5] [,6] [,7] [,8] [,9] [,10]
day "3/15" "3/15" "3/15" "3/15" "3/15" "3/15" "3/15" "3/15" "3/15" "3/15"
time "09:00" "09:15" "09:30" "09:45" "10:00" "10:15" "10:30" "10:45" "11:00" "11:15"
[,11] [,12] [,13] [,14] [,15] [,16] [,17] [,18] [,19] [,20]
day "3/15" "3/15" "3/16" "3/16" "3/16" "3/16" "3/16" "3/16" "3/16" "3/16"
time "11:30" "11:45" "09:00" "09:15" "09:30" "09:45" "10:00" "10:15" "10:30" "10:45"
[,21] [,22] [,23] [,24]
day "3/16" "3/16" "3/16" "3/16"
time "11:00" "11:15" "11:30" "11:45"
-
で,
こんなかんぢで時間割表生成 & CSV ファイル生成のコードを書いてみる.
# 口頭発表配列
table.ptime <- sapply(1:sum(n.t), get.ptime)
df.p <- data.frame(
day = table.ptime[1,],
time = table.ptime[2,]
)
m.p <- sapply(
1:n.u, function(i) {
unit <- list.u[[i]]
v.p <- rep(unit, table.g[unit])
k <- 0
col.p <- sapply(
1:sum(n.t), function(time) {
k <<- k + 1
v.p[k]
}
)
}
)
df.p <- cbind(df.p, m.p)
file <- "oral.csv"
cat("# output to", file, "...\n")
write.csv(df.p, file = file, row.names = FALSE)
-
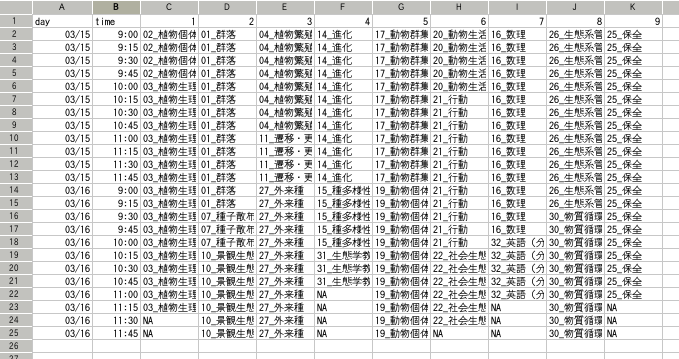
これを実行すると,
CSV ファイル
(注: 編成ボス点検用に Shift-JIS 化されてる)
が生成され,
ゑくせるもしくは
gnumeric
みたいなソフトウェアで開くと,
下の図のようになる.