
[ミミズ除去実験区]
苫小牧研究林は
なぜかしら日本国内でもかなりミミズ密度の高い森林らしい.
このミミズの働きを調べるため,
ミミズのぞいた実験区を防御壁で囲ってる様子.
かなりたいへんな作業だったそうで.

……
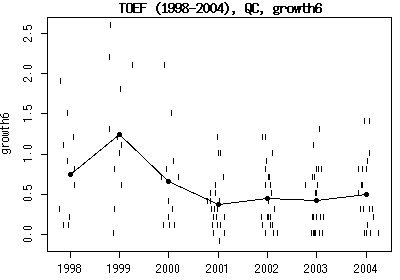
# AIC = 2210.97
# min AIC = 2071.37
# --- wv.mb set = 7/27 ---
# --- wv.ps set = 39/54 ---
temp.ps: best=16.0/width=6.0
ppfd: factor=0.02
vpd: factor=1.00
1998 1999 2000 2001 2002 2003 2004
26.65381 23.59560 22.93142 24.22508 25.75016 24.64961 25.81366
# AIC = 2218.24, np = 4+8 (scale.log const ps.prev mb6)
scale.log const ps.prev mb6
-0.53700289 1.22804230 0.01813444 0.15193820
# AIC = 2218.24
# min AIC = 2071.37
# --- wv.mb set = 7/27 ---
# --- wv.ps set = 40/54 ---
temp.ps: best=17.0/width=6.0
ppfd: factor=0.02
vpd: factor=1.00
1998 1999 2000 2001 2002 2003 2004
26.64290 23.88847 23.50196 25.19602 25.54211 23.64804 24.48687
……
てなかんぢで進行中.
27 × 54 = 1458 気象値セットしか調べてないのに,
たぶん 7 時間ぐらい必要.
次はこの「同時選抜の高速化」
をやらんといかんね
……
/etc/apt/sources.list
を変更して
sudo apt-get update && sudo apt-get dist-upgrade
すると,
えらくたくさんの rpm が入れかわってしまった.
X.Org
まわりとか.
glmmPQL()
に関する質問 (のひとつ) の回答を検討する
……
結果に Log-likelihood
とか出てまぎらわしいな.
しかも summary()
とると,
ご丁寧にも AIC & BIC 計算している.
ふーむ.
ともあれ返信.
glmmPQL()
は一般化線形モデルを quasi-likelihood (QL) ふうに
定式化して,
lme()
(in nlme)
に渡してるだけなんで計算とかは確実っぽく見える
(う? PQL の pennalized はどこにいった?).
そうか,
なんでパラメーター推定値に関して t 検定やるのか,
といった疑問については
nlme
方面を調べないとわからないわけだな.
いや,
最大 QL 推定値の分布ってわかってるのかしらん?
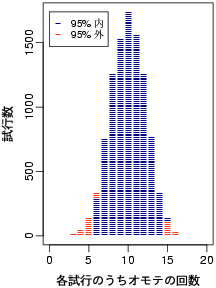
dbinom(0:20, 20, prob = 0.5))
……
試行回数が 1000 ぐらいだと,
ぜんぜん二項分布っぽくなくて面白い.
で,
10000 とかにするんだったらむしろ
barplot()
すべきだったかも,
と後になってから思いついたり.
jpeg()
出力と
jpeg2ps
連携わざでいくことに.
jpeg()
では日本語が出力されんか.
じゃ,
敵国語で,
と調べてみる
……
コインのオモテは obverse
(俗語では heads),
ウラは reverse (tails)
……
いや軸ラベル問題は LaTeX で片づけるか.


glm()
のような単純なやりかたは使えない.
しかしながら,
直径成長年変動の説明に使う代謝気象値・光合成気象値を
数千・数万の候補の中からしぼりこむには
(特定するわけではない),
glm()
みたいな計算で十分,
とわかった.
これでうまくいけば 7-8 時間かかっていた計算を 10-20 分ぐらいに
短縮できる.
glm(..., family = poisson)
はまずかったか?
glm(..., family = quasipoisson)
にしてみる.
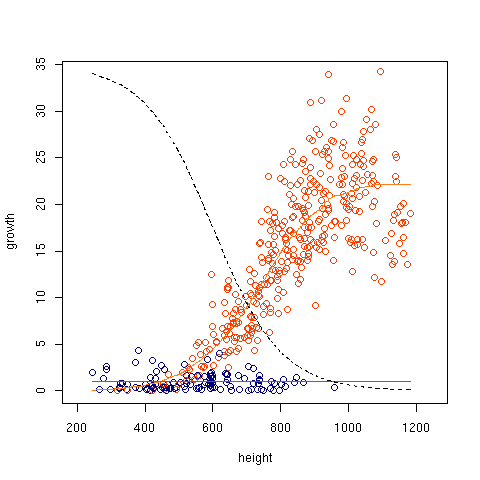
平均 vs 分散の関係はこんなふうだし
……
あれ?
ミズナラでは分散がだいたい平均に比例してるぞ?
quasipoisson
推定で使うのは擬似尤度だから
aic
は計算できん.
そこで
deviance
(これも擬似っぽいな)
見て選抜かけてみる
……
ダメだ.
ミズナラだけはどうしてもうまくいかん.
他の 5 樹種は完ペキなのに.