
[ホイコーロー定食 456 円]
原価 100 円ぐらいかな
……
安いのか高いのか
……
キャベツたくさん食った,
という食後感.
library(lme4)
の glmer()
とかダメ
……
少なくともかなり注意ぶかく使わないといけない.
version によって推定値とかぜんぜんちがう
stepAIC()
する窮余の一作として glm.nb()
をひさしぶりに使う
……
しかしこの組み合わせも NA な説明変数にはヨワい
(除去するしかない),
また glm.nb() は control = glm.control(maxit = 1000)
といった指定が必要不可欠



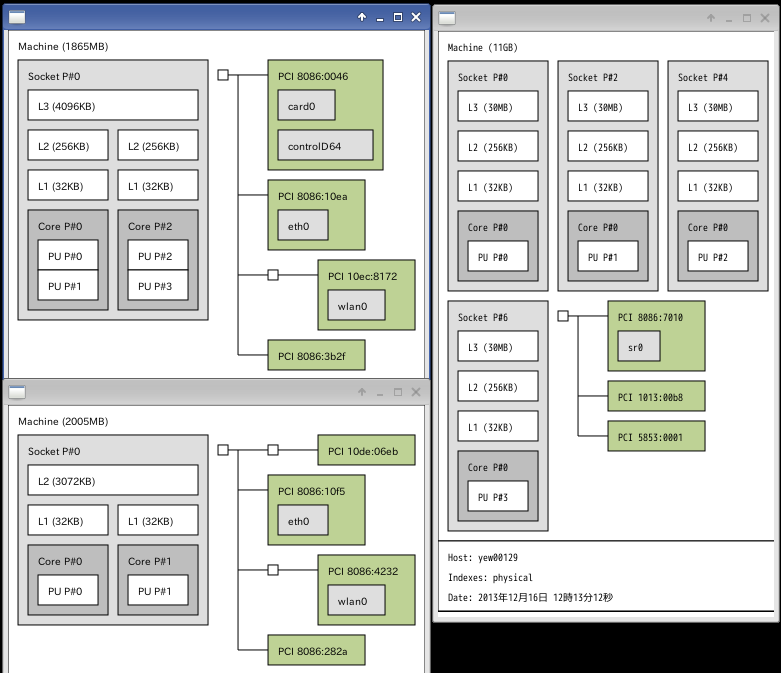
sudo apt-get install hwloc
して
hwloc-ls -p
するとこうなる.
ThinkPad X201s (左上),
Dell Latitude E6500 (左下),
北大くらうど
(右 …… すげー).
dixon2002()
関数は良さそうだな
……
nearest neighbor contingency table ってのがいいよね.
「統計量」としての分割表,
といったかんじで.
平均距離とかいった統計量よりよさげなかんじがする.


どうも私は植生学の考えかたとデータ解析手法が何かうさんくさいものだと考えているようで,例の野外実験の経過観察データを植生学的な発想・手法であつかうことに疑問をもっているという状況なのでしょう.
私自身は植生学は勉強したことがなく,露崎さんの大学院生たちがときどき植生学的な多変量解析とかをやるので,その手法あれこれ関連性や妥当性を検討してみたり,手法の背後にある「考えかた」について文献を少し読んでみたぐらいです.
とりあえず,私自身の理解としては「植生なヒトたちはいくつかのれ環境傾度 (連続的・一次元的で単純な尺度が前提?) の単純な組み合わせ (直交性みたいな) 植物群集の構成 (つまり群落構造) が決まると考えている」ので,ああいう多変量解析をやっているのだろうといったところです.釜野さんがいろいろな方法をあげていますが,といった疑問がつらつらとわいてくるんですよね.とりあえず,環境傾度の組み合わせ→群落構造というのあまり「実証」されていないような気もします.
- 多変量解析あれこれは本当に上のような考えかたにそったものなのか
- そもそも上のような考えかたは現代の生態学で妥当なものだろうか
さきほどのメイルで書かなかった,植生学的な多変量解析についてのいくつかのコメント,みたいなものを書いてみます.これまた私が本とかをぱらぱらながめただけの独学にもとづくもので,自分自身でソフトウェアを使ってみた経験もないような状態なのですが,以前から気になっていたので,ちょっとそのあたりを……
植生学的な多変量解析は基本的にどれも古いものが多く (つまり現在よりずーっと性能の悪い計算機で動作しなければならないという制約条件のもとで開発されたものです),現代的なデータ解析の考えかた (たとえばデータのばらつきみたいなものを考慮する,など) がうまく組みこまれていなくて,その結果として「データセットをちょっと変えると,結果ががらっとかわる」みたなことがおこりやすいかもしれません.
露崎さん雑談 (半分冗談?) のひとつとして「都合のいい ordination できなかったらどうするか? いくつかの調査地をぬいてみたりすると,それっぽくなったりすることもあるんだよね〜」といったハナシもあります (もちろん現代的な統計モデリングでもこういった種類の操作は不可能ではありませんが).
TWINSPAN と DCA には共通点があります.それはどちらも reciprocal averaging (RA) あるいはその別名 corresponding analysis (CA) による ordination をやっていることです (と小林本に書いてあるけど,ホントなのだろうか?).
TWINSPAN は,そのあと指標種 (私にはこのあたりよくわかりません) をにらみながら,さらに並び替えをやって,その後「分割」するわけですね.いっぽうで DCA というのは D こと detlended なる凶悪な操作をやって第 2 軸の方向に生じている「歪み」を強制的に無くしてしまうという……私はいろいろな数理的手法を勉強しましたが,これは現用されている方法の中では,かなりひどいものです……ともあれ,どちらも CA なる操作をしているので,理論的に考えると,似たような結果になっていても不思議ではありません.ただし TWINSPAN は最後にナゾの並び替えをやるのでその効果がよくわからない,あるいは,PC-ORD と R の library(vegan) では CA なんかの計算方法が違っていて,両者がうまく対応しないという可能性はあります.
nMDS は上とはまた異なる方法で,何らかの群集類似度で測られた plot 間の「距離」にうまく対応するように,plot ごとの多変量データをこねあげてつくった統計量を作りましょう,というものですよね.
この「対応」づけにおいて,nMDS の n こと non-parametric な方法で並べる,つまり私が前のメイルで書いた「尺度」の問題が気になるので,「plot ごとの値の大きさ」そのものではなく大小関係だけをみて序列化しましょう,といった方法です……これまた,私はぜんぜん使った経験がないので,良いのか悪いのかいまいちぴんとこないです.
たとえばlibrary(vegan)で nMDS をするときにはvegedist()関数で群集間距離を決めるようですが,この群集間距離みたいなものがいろいろ選べるようになっていますよね……距離計算の方式を変えると結果がどう変わるんだろうといったことも気になります.