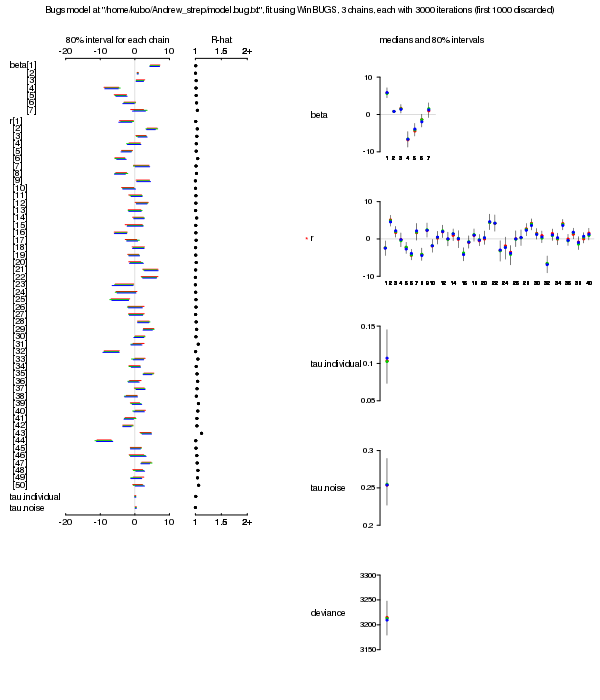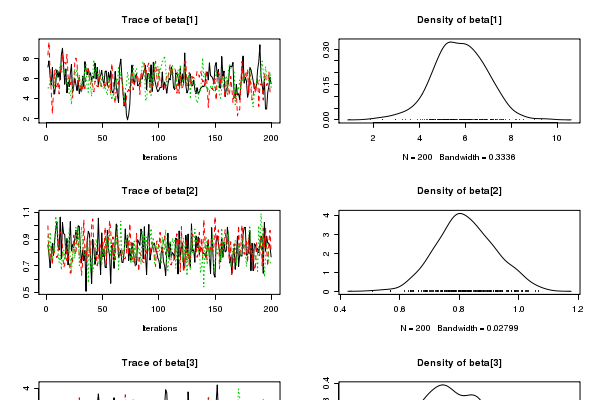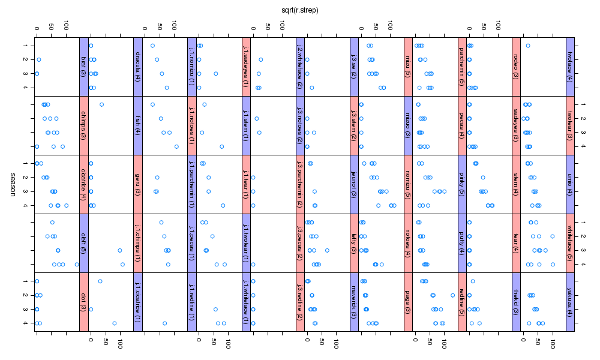
help(dbeta) などの
help file
をみると,
dbeta(x, shape1 = a, shape2 = b)
とパラメーターを指定すると
M <- a / (a + b)
V <- a * b/((a + b)^2 * (a + b + 1))
Q <- M * (1 - M) / V - 1
とすると),
a <- M * Q
b <- (1 - M) * Q
logit(M) <- b0 + b1 * x1 + b2 * x2 + ...
などとモデリングすればよい.
V <- M * (1 - M)
とか?
右辺を比例係数をかけてもいいのかもしれないけど.
具体的な計算方法ですが,このように構築した統計モデルを, ・R の optim() 関数で最尤推定する ・WinBUGS や JAGS を使って階層ベイズモデル化して,MCMC で事後分布を推 定する といった手段がありそうです.郷さんの問題のややこしさからいって,ベイズ モデル化が無難な手法かもしれません.上で書いたような遺伝子発現のモデリ ングはすでに誰かがやっているのではないかと思いますよ. なんとも中途半端なメモとなりましたが,解析の進展のヒントにでもなれば, ということで書いてみました.と書いて郷さんにメイル送信してみる. われながらいいかげんだなぁ …… ここで議論してるモデリング対象は DNA マイクロアレイ, この発現率なる割算値はだいじょうぶなのかね? といつも疑問に思ってしまうのだが ……