えーと,
てきとうに例題を作って
fit1 <- lmer(y ~ (1 | group), family = binomial, method = "Laplace", ...) # glmer オブジェクト
をやると
Generalized linear mixed model fit using Laplace
Formula: y ~ (1 | group)
Family: binomial(logit link)
AIC BIC logLik deviance
1385 1395 -690 1381
Random effects:
Groups Name Variance Std.Dev.
group (Intercept) 0.0353 0.188
number of obs: 1000, groups: group, 10
Estimated scale (compare to 1 ) 0.9976
Fixed effects:
Estimate Std. Error z value Pr(>|z|)
(Intercept) 0.108 0.087 1.24 0.22
となり,
さらに
mcmc1 <- mcmcsamp(fit1, n = 500) # library(coda) の mcmc オブジェクト
やってから
(library(coda)
も忘れず実行)
summary(mcmc1)
やると
Iterations = 1:500
Thinning interval = 1
Number of chains = 1
Sample size per chain = 500
1. Empirical mean and standard deviation for each variable,
plus standard error of the mean:
Mean SD Naive SE Time-series SE
(Intercept) 0.109 0.0999 0.00447 0.00528
log(grop.(In)) -3.391 1.1070 0.04951 0.14390
deviance 1385.842 6.2637 0.28012 0.47325
2. Quantiles for each variable:
2.5% 25% 50% 75% 97.5%
(Intercept) -0.084 0.0474 0.104 0.169 0.318
log(grop.(In)) -6.022 -3.9808 -3.269 -2.629 -1.521
deviance 1375.724 1381.4101 1385.200 1389.406 1399.566
で fit1 と summary(mcmc1)
を比べてよーやくわかったんだけど,
MCMC 計算の結果に含まれてる
log(grop.(In))
ってのは group の random effects
(つまり group 差)
あらわす事後分布の分散の log
になってるわけね.
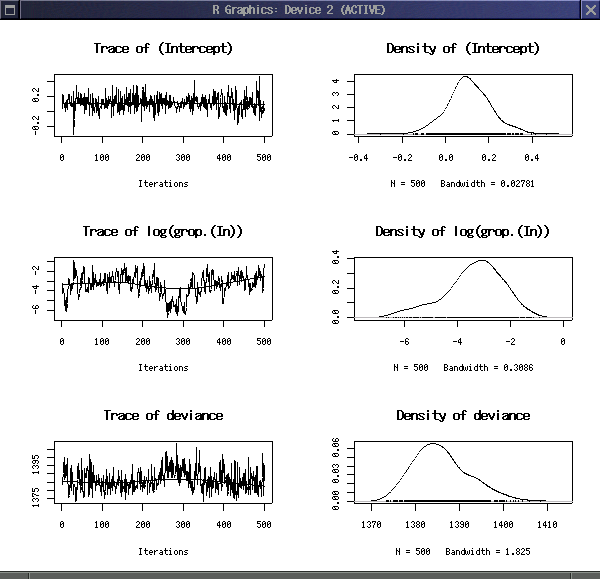
plot(mcmc1)
すると,
いつものごときこういう図になるわけで.