[1 parameter model]
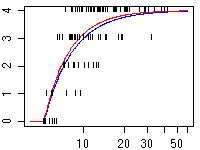
もっとも簡単な確率論的開花モデル.
二項分布なんで見た目の「ずれ」は大きい.
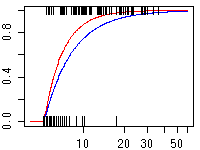
青線が ``|'' を吐きだしたモデルで,
赤線がそれを推定してみたもの.
optim() から評価するたびに,
parameter mapping という操作が必要になる.
いままではそれを
R
の中でやってから,
# p -> mp (mapped paramters) matrix.map <- rbind( c(1:length(map.parameters))[map.parameters == 1], parameters ) mp <- map.parameters for (r in data.frame(matrix.map)) mp[r[1]] <- as.numeric(r[2])C で書いた拡張モジュールにわたしていた.
growth6 のみの推定;
growth6 & growth7
同時推定だと 230-240 秒ぐらい).
さーて,
parameter mapping in C 版だと
……
16-17 秒というところか.
なかなかいいぞ.
意外と速くなった.
いままで 6 時間ぐらいかかってた計算が
4 時間ぐらいで片づくようになったわけで.
R
の optim()
ではいろんな計算法が指定可能なんだけど,
頑健な (しかし遅い) Nelder-Mead 法はこういう
1 parameter 推定にはむいていない.
そこで BFGS 法を使った.
これは勾配関数を指定する必要あり.

rbinom(100, 4, prob.flower(DBH))
で生成したパターンであるのに,
思ってたよりばらつきがでかいように見える.
見た目で overdispersion かどうか判定するのは危険だな.