

library(MCMCglmm)
を使った GLMM のパラメーターの MCMC サンプリングを複数回
(3 回とか)
を独立にやる
library(R2WinBUGS)
が定義する bugs
オブジェクトに変換する
bugs オブジェクトに変換すれば,
Gelman-Rubin の R-hat の計算とか,
あるいはいろいろな図示がラクになる
……
といったもくろみ.
lmer() なんかは使うのヤメて,
MCMCglmm() を!」
とススめているんだけど
(皆さん WinBUGS とか JAGS なんかを使用すると,
なぜかしら健康をいささか損なうおそれがある
……
といった迷信にとりつかれているようなので)
……
いつものごとく「あーいえば,こーいう」といいますか,
「これってどうやってサンプリング数とか決めたらいいんですか?!」
といかにも院生じみた質問・詰問ばかりが返ってくる日々に,
はなはだしく疲労困憊してしまったため.
library(MCMCglmm)
burnin <- 1000
thin <- 50
nitt <- 1000 * thin + burnin
n.chain <- 3
list.MCMCglmm <- lapply( # ここは並列化したほうが良い
1:n.chain,
function(chain) MCMCglmm(
cbind(peak_no, flower_no - peak_no) ~ log(stem_Do) * sex,
random =~ stem_id + ind_id,
data = d, family = "multinomial2",
pr = TRUE, # !!! これはめんどうのもと (後述) !!!
nitt = nitt, thin = thin, burnin = burnin
)
)
source("merge.MCMCglmm.R") # 関数定義ファイルのよみこみ
post.bugs <- merge.MCMCglmm( # 変換
list.MCMCglmm,
n.iter = nitt, n.burnin = burnin, n.thin = thin
)
以上のように R 内で実行して,
いつものごとく plot(post.bugs)
などとやると,
以下のように図示されたりする.
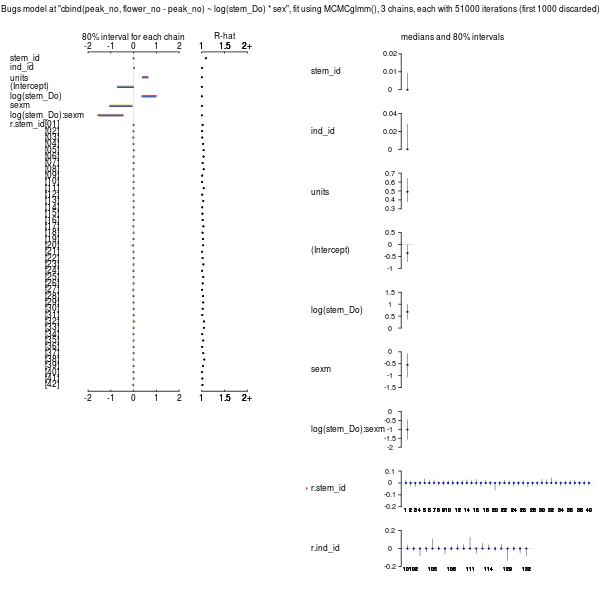
print(post.bugs, digits.summary = 3)
すると以下のように出力される.
Inference for Bugs model at "cbind(peak_no, flower_no - peak_no) ~ log(stem_Do) * sex", fit using MCMCglmm(),
3 chains, each with 51000 iterations (first 1000 discarded), n.thin = 50
n.sims = 3000 iterations saved
mean sd 2.5% 25% 50% 75% 97.5% Rhat n.eff
stem_id 0.005 0.019 0.000 0.000 0.000 0.000 0.063 1.089 28
ind_id 0.012 0.042 0.000 0.000 0.000 0.000 0.139 1.008 280
units 0.502 0.102 0.332 0.429 0.491 0.565 0.727 1.001 3000
(Intercept) -0.357 0.288 -0.930 -0.549 -0.350 -0.164 0.203 1.001 3000
log(stem_Do) 0.680 0.257 0.161 0.508 0.678 0.853 1.188 1.001 2600
sexm -0.551 0.386 -1.310 -0.810 -0.549 -0.300 0.201 1.001 3000
log(stem_Do):sexm -1.012 0.438 -1.862 -1.303 -1.008 -0.710 -0.167 1.001 3000
r.stem_id[01] 0.007 0.077 -0.105 0.000 0.000 0.001 0.193 1.018 3000
r.stem_id[02] -0.003 0.070 -0.149 -0.001 0.000 0.000 0.118 1.006 3000
r.stem_id[03] -0.010 0.079 -0.217 -0.001 0.000 0.000 0.086 1.010 2800
r.stem_id[04] -0.004 0.064 -0.143 -0.001 0.000 0.000 0.106 1.029 1500
r.stem_id[05] 0.010 0.074 -0.091 0.000 0.000 0.001 0.213 1.041 470
r.stem_id[06] 0.003 0.067 -0.122 0.000 0.000 0.001 0.174 1.038 1200
r.stem_id[07] 0.009 0.074 -0.079 0.000 0.000 0.001 0.172 1.018 3000
r.stem_id[08] -0.004 0.066 -0.168 -0.001 0.000 0.000 0.114 1.042 1400
r.stem_id[09] 0.000 0.067 -0.124 0.000 0.000 0.000 0.128 1.010 2600
r.stem_id[10] 0.003 0.070 -0.109 0.000 0.000 0.000 0.145 1.007 3000
r.stem_id[11] 0.004 0.079 -0.118 0.000 0.000 0.001 0.154 1.041 670
r.stem_id[12] -0.004 0.071 -0.162 -0.001 0.000 0.000 0.114 1.011 3000
r.stem_id[13] 0.002 0.069 -0.126 0.000 0.000 0.001 0.142 1.033 1700
(略)
MCMCglmm → bugs
オブジェクト変換で面倒なのは,
pr = TRUE と指定したときに MCMC サンプリングされる,
いわゆる random effects なパラメーターの名前を書き換えねばならぬところ.
MCMCglmm オブジェクト
(注: pr = TRUE と指定して生成したもの)
が x であるとすると,
x$Sol という行列には
"stem_id.i01",
"stem_id.i02",
"stem_id.i03", ...
といった列名 (colnames)
で MCMC サンプルが格納されているので,
これを
sub("(.+_id)(\\.)([A-Za-z_]*)([0-9]+)", "r.\\1[\\4]", ...)
といったかんじの
呪術的
というしかない R 正規表現で置換した.
r. という文字列をつけたのは,
これをやっとかなないと plot(post.bugs)
がエラーになるため
……
さらに,
これらの添字が 1 から始まる通し番号でないときには,
plot(post.bugs) の左パネルには正しく図示されないみたいだ.
上の図でも ind_id は示されていない
(右パネル一番下には図示されているが).
mosaicplot()
とか
plot.table(),
spineplot() とかかな.
library(lattice)
で 検索
すると barchart.table()
(総称関数 barchart)
……
まだインストールしてないけど,
library(vcd)
にはいろいろな関数が用意されてるみたいだ.
vcd とは visualizing categorical data とのこと.
demo(package = "vcd")
もいろいろ準備されてるな.
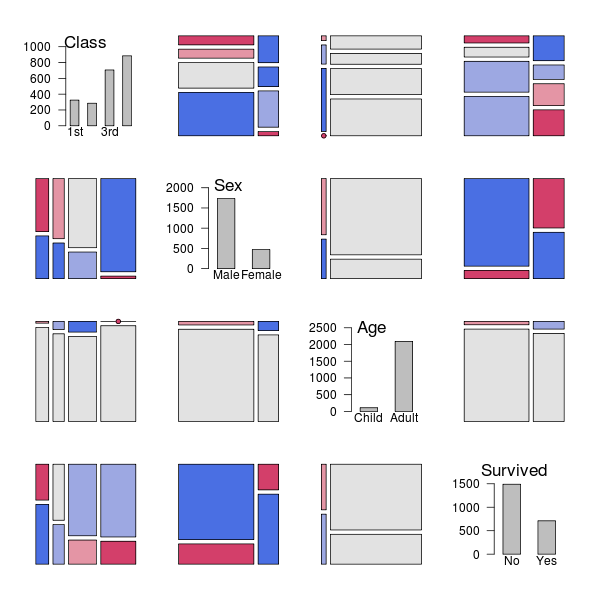
これはタイタニック号遭難事件時の,
客室等級・男女別・年齢ごとの生死.
library(bipartite)
の plotweb()
とか.
これはすごい.
以前,
苫小牧にいた平尾君も同様の関数を作ってはいたのだが
(ぎょーむ日誌 2005-03-17).
