
[とりあえずらくがき & はりつけ]
いきなり文章とかがすらすら出るはずもなく,
いろいろとらくがきしたり,
そのへんにぶらさげてみたり
……

glm()
であつかっていたポアソン回帰 → 無情報事前分布なベイズモデル化 &
WinBUGS の導入
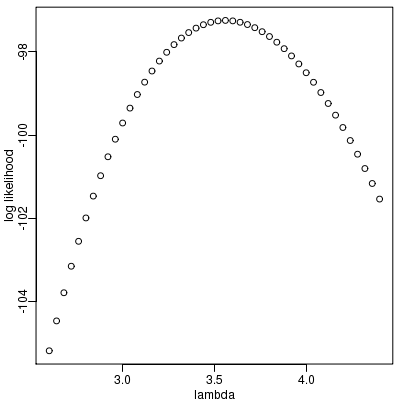
glm() で十分な問題
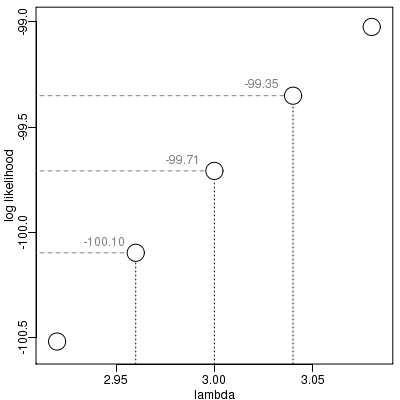
p12 <- 0.5 # prob(i -> i.next)
p21 <- 0.5 # prob(i.next -> i)
if (i == i.min | i == i.max) {
p12 <- 1
} else if (i == i.min + 1 & i.next == i.min) {
p21 <- 1
} else if (i == i.max - 1 & i.next == i.max) {
p21 <- 1
}
p <- exp(vlogL[i.next] + log(p21) - vlogL[i] - log(p12))
で,
サンプリングさせてみると
……
やっぱ WinBUGS とかに比べると格段に収束がわるいね.
下の例は 100 step とばしに 1000 回サンプリングした結果なのだが.
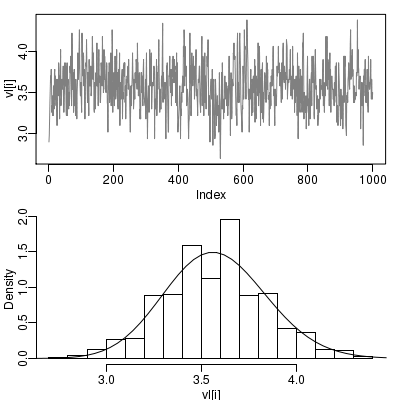
plot(density(事後分布標本))
すると「なめらかに & もっともらしく」見えるようになってしまうんだよね
……