[隠し PC 復旧]
お茶区画の EP なところにおいてる desktop PC.
なんかいきなり起動しなくなって,
当時は岩波本ですまーちの佳境で (つまり計算用 PC いらん),
節電があーだこーだと言われていたときなので,
修理しないで放置していた.
今日しらべてみたらファイルシステムがちょっとヤラれてただけだったので,
fsck.ext3
で修復.

fsck.ext3
で修復.
/proc/cpuinfo
を調べてみると,
cpu family : 15 model : 2 model name : Intel(R) Pentium(R) 4 CPU 2.40GHz stepping : 7 cpu MHz : 2398.794 cache size : 512 KBほらほらクロック数は 2.4GHz で, ThinkPad は
cpu family : 6 model : 37 model name : Intel(R) Core(TM) i7 CPU L 640 @ 2.13GHz stepping : 5 cpu MHz : 2134.000 cache size : 4096 KB2.1GHz …… うーむ, しかし cache size とかぜんぜんちがうし, やはり Pentium 4 と Core i7 のあいだには, クロック数みたいな単純な数値では説明できない 何か隔絶した CPU 設計の進化があって, 計算速度にこれほどの差が生じてしまったのだろうか?
library(foreach)
library(doMC)
registerDoMC(cores = 4) # use 4 cores
list.bugs <- foreach(job = 1:3) %dopar% {
wd <- sprintf("tmp%i", job) # i.e. "tmp1", "tmp2", "tmp3"
if (!any(dir() %in% wd)) dir.create(wd)
call.bugs(
file = "model.bug.txt",
n.chains = 1,
debug = F,
bugs.seed = round(runif(1, -10^6, 10^6)), # random seed
working.directory = wd,
n.iter = 30000, n.burnin = 5000, n.thin = 50
)
}
要点は
bugs.seed
で WinBUGS の「乱数のたね」を
bugs.seed
は当初まったく気づかなくて,
この日の午後は悪夢のごとき bugs
クラスのコード読み地獄にはまりこんでしまった
……
ユーザ システム 経過
123.99 0.40 129.07
並列化すると
ユーザ システム 経過
63.287 0.288 69.835
ユーザ システム 経過
74.228 0.212 81.263
ユーザ システム 経過
76.764 0.296 86.264
こうだから 124 秒ぐらいかかってたのが 77 秒ぐらいで終了ということ.
まあ,
これはゐんばぐすの起動とかコードのコンパイルや初期設定に時間とられているんだろう
……
などと思っていたんだけど,
あとでもっと長い計算で比較してみても,
やはり 3 倍ではなく
2 倍
ぐらいの高速化にとどまる
(1 core だと 4.5 時間かかるはずの計算,
4 core だと 2.3 時間ぐらいかかった)
……
ようで.
いろいろおーばーへっどとかあるのかな?
まあ 2 倍でもじゅうぶんありがたいけどね.
list.bugs
というところに結果が格納されるんだけど,
これは n.chains = 1 の bugs
3 個の list になっている.
これではいろいろと不便なので
……
n.chains = 1 な bugs 3 個から,
ひとつの n.chains = 3 な bugs
をねつぞうしてやろうという問題なのだが
……
よくわからなくて,
あちこち走りまわってカベにあたまうちつけてひっくりかえって
(呪われぎみな bugs オブジェクトの構造は
こちらで解説してます)
……
merge.bugs <- function(list.bugs)
{
b1 <- list.bugs[[1]]
m1 <- b1$sims.matrix
mall <- matrix(numeric(0), 0, ncol(m1))
n.chains <- length(list.bugs)
for (i in 1:n.chains) {
mall <- rbind(mall, list.bugs[[i]]$sims.matrix)
}
sims.array <- array(mall, dim = c(nrow(m1), n.chains, ncol(m1)))
dimnames(sims.array) <- list(NULL, NULL, colnames(m1))
as.bugs.array(
sims.array = sims.array,
model.file = b1$model.file,
program = b1$program,
DIC = FALSE,
DICOutput = NULL,
n.iter = b1$n.iter,
n.burnin = b1$n.burnin,
n.thin = b1$n.thin
)
}
ようするに,
各 bugs オブジェクトから結果を matrix
のカタチでとりだし,
それを (サンプル数, chain 数, パラメーター数)
という 3 次元の array に整形して,
あとは library(R2WinBUGS) の as.bugs.array()
に投げてしまうという策.
bugs
おぶぢぇくとに変換すれば plot(bugs) とかできるでしょう.
list.bugs
内にある 3 つの bugs オブジェクトを統合して,
ひとつの n.chains = 3 な bugs
がえられる
……
post.bugs <- merge.bugs(list.bugs)
……
なぜだ?
R-hat がぴったり 1 になっている?
bugs.seed
問題で
……
このときは bugs.seed
を指定してなかったんだよね.
そうすると,
(user manual にも書いてないんだけど)
seed が何か特定の値 (たぶん 0 か 1) にセットされてしまう.
そうすると,
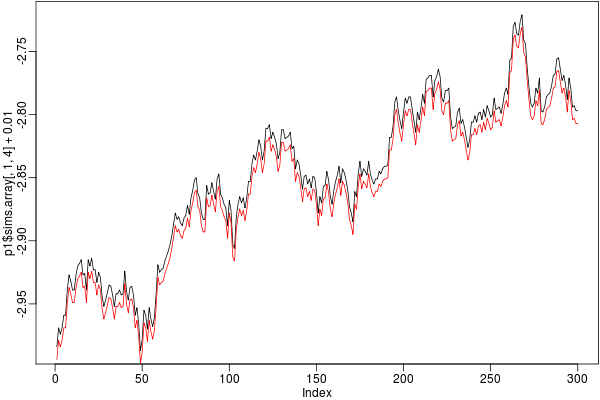
n.chains = 1 の MCMC サンプリングを 3 回実施しても,
下の図のように,
完全に同じものしか得られないわけで
……
下の図は,わかりやすくするため,ちょっと上下にずらしてます.
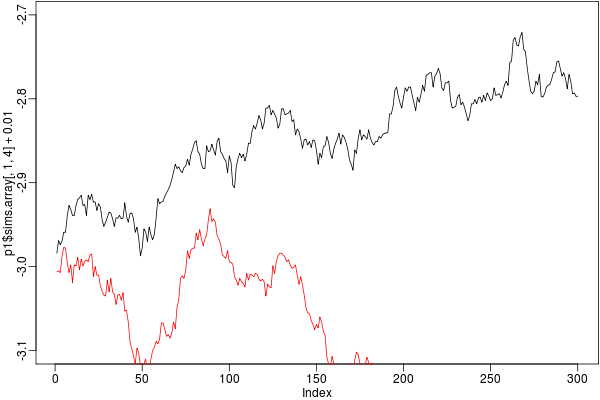
bugs.seed
を設定する.
すると,
このようになる.
n.chains = 1
の MCMC サンプリングのプロセスたちを走らせるときには,
それぞれ異なる bugs.seed を指定すること.
ホントは初期値も乱数とかで与えて変えるべきなんだけど,
現在の R2WBwrapper.R ではまだそれはできない
……
初期値のあつかいはだいたいわかってきたので,
そのうちに改造します.
ユーザ システム 経過
6867.62 9.56 6919.35
ユーザ システム 経過
8389.471 2.392 8435.749
ユーザ システム 経過
8408.1 5.4 8463.8
ではでは,
post.bugs <- merge.bugs(list.bugs)
してと
……
えーい,
もっと長くサンプリングしないとダメか.