
[R による医療統計学]
とても良い教科書.
「医療統計学」と書かれているけれど,
どの分野の人がみても参考になる.
生態系機能学総論(モデル中心)後期前半 月・木4時限 この講義では、生態系の機能を解析する手法として現在用いられている数理モ デル、統計モデルに関して基礎的な知識を概説することを目的とする。まず、 前半では動態を記述する数理モデルについて学び、生物の行動や形質の進化に ついて議論する数理モデルについて学ぶ.また、後半では、統計モデルの基礎 となる考え方や最尤法について学び、その上で一般化線形モデルやより高度な 統計モデルについて学ぶ。 (1-7 担当: 高田) 1. 数理モデルの概説 2. 動態編1(基礎モデル) 3. 動態編2(推移行列モデル) 4. 動態編3(群集モデル) 5. 進化編1(最適戦略モデル) 6. 進化編2(ESSモデル) 7. 進化編3(共進化モデル) (8-14 担当: 久保) 8. 生態学データ解析の統計モデリングとは? 9. さまざまな確率分布と最尤推定 10. 一般化線形モデル (GLM) 1 11. 一般化線形モデル (GLM) 2 12. 検定とモデル選択 13. 一般化線形混合モデル (GLMM) 14. 階層ベイズモデル

plot(glm(...))
ってあまり意味ないんじゃないかな.
glm()
はほぼ正確な推定値を計算してくれる.
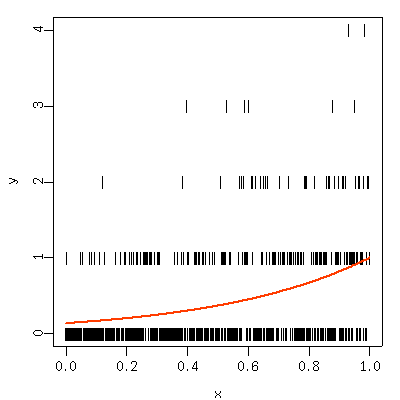
fit <- glm(y ~ x, family = poisson)
して
plot(fit)
するとどうなるか,
というと下のように何だか意味不明ぎみというか奇怪な Q-Q plot
出すんだよね.
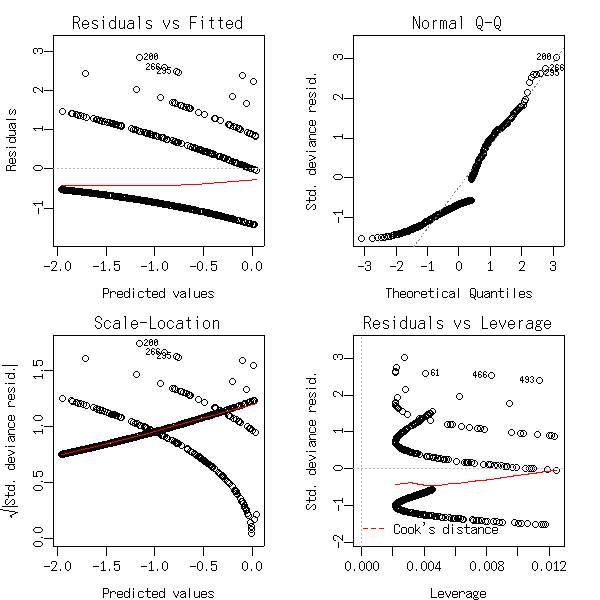
glm()
の出力オブジェクト
(glm オブジェクト)
でこんな図が書けるかといえば,
> class(fit) [1] "glm" "lm"とやればわかるよーに,
glm オブジェクト
は
lm オブジェクト
(正規分布の線形モデルの推定結果)
を継承して定義されたものであり,
glm(fit)
とすると
lm オブジェクト
の plot() メソッドである
plot.lm()
が呼ばれている,
というだけのことなんだよね
……
いやはや,
ヒトさわがせな.