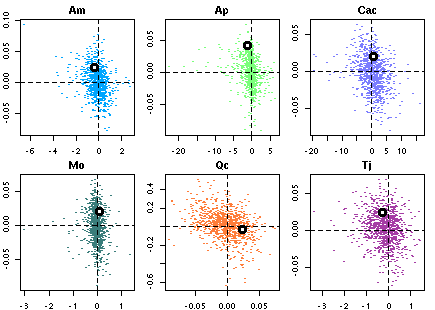
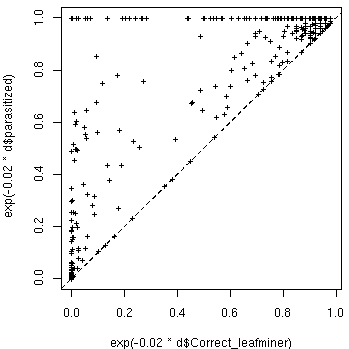
これの具体的な関数型としては f(d) = exp(-0.02 * d) というような密度の減少関数です…… と書いたところで, ちょい失敗をやったことに気づきました. d は現在のところ寄生された host 密度 (樹冠全体推定数) にしてますが, おそらくこれは host 全体にしたほうがマシかもしれません.なる修正の計算結果だが …… 結果がひっくりかえってしまった (しかしこの数時間後にさらにひどい状況になる, それを知るものはまだ人類のだれもが知らないことであった). こんどは apparent competition アリ, の方向ではないか (しかもミズナラ以外は同樹種間の影響が少ない) ……

> summary(d$Sp) Am Ap Cac Mk Mo Qc Tj 94 70 68 15 40 163 57が反映されてない, というのが発見の発端だったんだけど. うーむ. 一週間以上きづかなかった. ただちに修正. 計算はゼロからやりなおし …… といっても今まで使いものになりそうな結果が得られていないのは, まあ不幸中のさいわい, とでも理解すればいいのかもしれんが.
try()
で強引に計算してた理由はコレか
……
unless
がない.
しかしながら無限るーぷ repeat
はある.
ということで,
repeat {
density.randomized <- randomization(...)
fit <- glm(...) # MPLE 計算
if (fit$converged == TRUE) break # 終了
cat("ERROR: failure in conversion; retrying ...\n") # やりなおし
}
としてみる.
