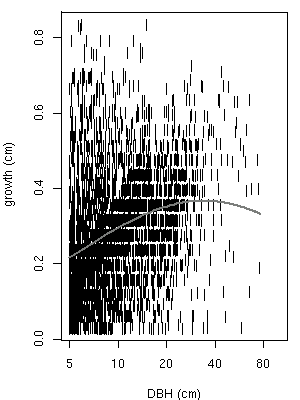
[いまいちアヤしい]
直径成長の分布.
まあ,
全 159 樹種こみだから,
なのかもしれないけど.
直径のばらつきはガンマ分布を仮定.
平均は exp(線形予測子).
offset 項として観測間隔を指定
(というのはちょっとイヤなんだけど,
割り算値とか使うよりはマシなんで).
分散は平均の定数倍
(このデータでは分散は平均より小さい).
よって,
glm()
とか使えない.
こういう場合は尤度関数を自分で書き,
optim()
など使って最尤推定値を計算する.